[1] 40Gap Term 2024 春
目的
本授業の目的はRを使うことによって
フローサイトメーターのデータを解析する
ことである
- Rの基本的な操作
- データ処理
- データの可視化
リンク
Part I: R入門
なぜコードを使うのか?
- 解析を再現可能にするため
再現性とは?
- 他の人(将来の自分を含めて)があなたの解析を
再度行って、同じ結果を得ることができること
なぜR?
- タダ
- 柔軟性
- 広く生態学に使われている
- コミュニティー
TokyoR
- 日本で最も大きなRユーザーのコミュニティー
- 定期的にミーテアップ
- slackで初心者(とそれ以上)の質問に答える
- https://github.com/tokyor/r-wakalangから誰も参加可能

RとRStudio
- Rとは「R Project for Statistical Computing」という組織が提供しているオープンソース(タダの)プログラミング言語
RとRStudio
- RStudioとはPosit社が提供しているIDE(Integrated Development Environment)
- 基本的な機能はタダ
- 会社向けの機能は有料
- Rのコードを書く・実行する最適なソフト
資料
R for Data Science
- 英語版はオンラインで無料 https://r4ds.had.co.nz/
- 日本語版はアマゾンなどから

解析の準備:プロジェクトを作る
File → New Project → New Directory → New Project
名前と置く場所を選択する。名前は
flowcyt-analysisにしよう。場所は自分が後で探しやすいところにしましょう(デスクトップがおすすめ)

プロジェクトを作る
RStudioが再起動されて、新しいプロジェクトに入る
右下の「Files」パネルに以下のファイルが入っていることを確認:
flowcyt-analysis.Rproj.gitignore(gitを使っている場合のみ)

.Rprojファイルについて
flowcyt-analysis.RprojはRStudioの設定を保存するためのファイルで、基本的には手でイジる必要はないでも、このプロジェクトを開くためには便利
- 一旦RStudioを閉じて、
flowcyt-analysis.Rprojのアイコンをダブルクリックすると、またプロジェクトに入ることが出来る。
- 一旦RStudioを閉じて、
データを置く場所を作る
- New Folderボタン(右下のパネル)をクリック→
data_rawというフォルダーを作る

データを置く場所を作る
- Teamsで共有した以下のファイルをダウンロードして、
data_rawに置きましょうitadori_flowcyt.xlsx

データを置く場所を作る
data_rawフォルダーを読み込み専用にしよう- 生データを手でいじらないのが鉄則
- Rに読み込んだ上でRの中で操りましょう
他のフォルダーを作る(割愛)
続いて、同じように他の以下のフォルダーも作る:
data_raw/- 生データを置く場所。生データは絶対にいじらない(読み込み専用)
data/- コードによって整えられたデータを置く場所。使い捨てのつもりで良い。
results/- コードによって得られた結果を置く場所。これも使い捨て。
生データとコードがあればいつでも結果は得られる(再現できる)
RStudioのデフォルト設定を
変えましょう
前のセッションのデータが残っていることは
再現的にダメ!
Tools → Global Options → General → Workspace
- “Restore .RData into workspace at startup” → 外す
- “Save workspace to .RData on exit:” → “Never”
RStudioのデフォルト設定を
変えましょう
前のセッションのデータが残っていることは再現の観点
からダメ!

Rを計算機として使う
- RStudioのコンソール(左下のパネル)にカーソルを置いて、簡単な計算をしてみましょう:
- おめでとうございます!Rプログラミングができました!
オブジェクト(変数)について
- 次に、Rの中のデータを変数(Rでは、「オブジェクト Object」と呼ぶ)として保存しよう:
オブジェクト(変数)について
これだけでは何も返ってこない。
変数の内容を確認するには、コンソールにその変数の名前を打てば良い:
関数とは
何かの値(インプット)を受けて、処理して、計算結果(アウトプット)を返すもの
関数とは
関数の書き方:
- 関数名(引数)
例えば
関数とは
関数の使い方を確認したい時は?関数名と打って、ヘルプファイルを参照すれば良い
スクリプト
- 今まではRに直接にコマンドを出していたが、これを毎回繰り返すのが面倒なので、コマンドをスクリプト(テキストファイル)に保存するのがおすすめ
- Rのスクリプトは
.Rもしくは.rの拡張子を使う
- Rのスクリプトは
- RStudioで新しいスクリプトを作成する方法
- File → New File → RScript
スクリプト
- 二つのスクリプトを作りましょう:
practice.R- 練習用(メモ書き)
ploidy-analysis.R- 解析用
パッケージとは
- Rパッケージとは、Rが出来ることを大幅に増やす「追加」のソフト(関数のまとまり)
- 現在、2万近くのパッケージがある!
- 今回は
xlsxファイルを読み込むためのパッケージ、そして他のデータ解析をやりやすくするパッケージをインストールする
パッケージのインストール
Rパッケージのインストールを行うには、install.packages()を使う:
一回インストールしたら、次回からはしなくて良いので、これはスクリプトに書かない
パッケージのロード
- パッケージをインストールしたら、使える状態するにはロードすることが必要
- インストールは一回でいい
- ロードは毎回必要
library()関数でロードする:
データの読み込み
今回のデータは
.xlsxなので、readxlパッケージのread_excel()関数を使う。このコードを
flowcyt-analysis.Rとして保存してください:
データの読み込み
flow_cyt_data_rawを打つと、読み込まれたデータが表示され、基本情報(列と行の数など)が表示される:
# A tibble: 414 × 12
通し番号 登録番号 種名 採取日 採取者 採取地 GPS 倍数性...8 トマト数値
<dbl> <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <dbl>
1 1 33-1 <NA> 20220627 上原 <NA> 35.6… 3.0 3975.
2 2 34-1 <NA> 20220628 上原 <NA> 35.2… 3.0 5452.
3 3 34-2 <NA> 20220628 上原 <NA> 35.2… 3.0 5496.
4 4 35-1 <NA> 20220720 上原 <NA> 36.5… 3.0 5050.
5 5 36-1 <NA> 20220720 上原 〒371-01… 36.5… 不明(2倍… 5896.
6 6 36-2 <NA> 20220720 上原 〒371-01… 36.5… 不明(2倍… 5042.
7 7 36-3 <NA> 20220720 上原 〒371-01… 36.5… 不明(2倍… 4896.
8 8 37-1 <NA> 20220720 上原 <NA> 36.5… <NA> NA
9 9 37-2 <NA> 20220720 上原 <NA> 36.5… <NA> NA
10 10 37-3 <NA> 20220720 上原 <NA> 36.5… <NA> NA
# ℹ 404 more rows
# ℹ 3 more variables: サンプル数値 <dbl>, `2C` <dbl>, 倍数性...12 <dbl>データの読み込み
Environmentパネルのflow_cyt_data_rawをクリックすると、エクセルのような画面が出てくる

データを整える
- 行の名前を確かめてください
- 文字化けを防ぐために、なるべくローマ字表記がいい。ローマ字に変えよう
データを整える
以下のコードを試してください:
# A tibble: 414 × 3
id_num tomato_peak sample_peak
<dbl> <dbl> <dbl>
1 1 3975. 6848.
2 2 5452. 8089.
3 3 5496. 7835.
4 4 5050. 8093.
5 5 5896. 0
6 6 5042. 0
7 7 4896. 0
8 8 NA NA
9 9 NA NA
10 10 NA NA
# ℹ 404 more rowsデータを整える
行名がスッキリしたでしょう?
でも、どうやってこのデータのバージョンの保存ができるのでしょうか?
データの(Rの中での)保存
データの(Rの中での)保存
大事:
<-を使わないと計算結果が保存されない同じ名前で保存すると(注意なしで)前のデータが上書きされる
コードがあれば、計算結果を再現できるので、上書き・消去は気にしなくて良い
列の順を並び変える
arrange(data, column)- デフォルトで小さいから大きい順
列の順を並び変える
- デフォルトで小さい順(あるいは、Aの方)から並ぶ
- 大きい方(あるいは、Zの方)から並べたい時は
desc()を使う
# A tibble: 414 × 3
id_num tomato_peak sample_peak
<dbl> <dbl> <dbl>
1 370 7684. 32739.
2 371 7573. 32208.
3 375 7565. 31624.
4 205 7641. 31514.
5 138 7473. 31456.
6 394 7387. 31344.
7 359 7484. 31308.
8 395 7428. 31116.
9 142 7258. 31080.
10 290 7346. 31050.
# ℹ 404 more rows行を抜き出す
select(data, column)で行を抜き出す
行を抜き出す
select(data, column1, column2)で行を抜き出す- 複数の行を選ぶこともできる
行を抜き出す
- 全く別な方法もある:
$$を使うと、行の中身が返ってくる(データフレームではなく)
[1] 6847.69 8089.15 7834.95 8093.26 0.00 0.00 0.00 NA
[9] NA NA 9751.62 NA NA NA 8487.68 NA
[17] NA 10504.04 NA 10224.87 NA NA 11933.59 NA
[25] NA NA 11557.79 NA 9873.34 NA NA NA
[33] NA 23431.05 NA 11624.41 7108.45 NA NA 6805.19
[41] 7978.19 10870.25 23204.67 20443.44 18342.00 8445.05 9053.59 NA
[49] NA NA 13038.58 10920.38 NA NA 10908.05 10250.30
[57] NA NA NA NA 21065.42 NA 10885.63 NA
[65] NA NA 7088.15 6916.84 NA NA 14592.24 NA
[73] 15607.39 NA 15648.77 NA 14836.38 NA NA 15358.21
[81] 14907.62 NA NA 15501.19 NA NA NA 15110.68
[89] NA NA 15846.25 NA NA NA 15468.13 NA
[97] NA 15654.54 NA NA 15549.21 NA 14993.32 14971.91
[105] NA NA 15766.92 NA NA NA 15882.21 NA
[113] NA 14764.57 15459.79 NA NA 15618.64 NA NA
[121] 14156.51 NA NA 15375.92 NA 15646.18 NA NA
[129] 15637.52 NA NA 15260.94 NA 14066.98 14332.10 NA
[137] NA 31456.15 NA NA 21237.13 31079.73 NA NA
[145] 30599.29 NA NA NA 30330.38 NA NA NA
[153] 30265.68 NA NA 29467.32 NA NA 30784.13 NA
[161] NA 26668.67 NA 30498.02 NA NA 28809.97 NA
[169] NA 30229.66 NA NA 21723.27 NA 30892.73 NA
[177] NA 30247.34 NA 30696.10 NA 29625.03 NA NA
[185] 30739.64 NA NA 16010.41 NA NA NA NA
[193] NA NA 16072.27 14980.46 15257.21 14641.25 31008.20 NA
[201] 30779.92 30739.18 NA 30537.03 31514.44 NA NA 16788.72
[209] NA NA NA NA NA NA NA 15888.38
[217] NA NA 28150.10 NA NA NA 30494.79 28324.28
[225] NA 28271.56 29866.12 28882.77 NA NA NA 15782.04
[233] 16044.87 30696.23 29784.86 15445.38 NA NA NA NA
[241] 9657.13 15776.70 NA 10581.02 11483.13 17078.50 10654.86 NA
[249] NA 18386.86 NA NA 17506.39 NA 14332.03 12550.84
[257] NA NA NA NA 9357.72 NA NA 9823.48
[265] NA NA NA 22868.36 NA NA NA 9620.30
[273] 8446.23 8719.02 11488.66 9777.74 NA 8166.90 10367.22 13412.04
[281] 10608.76 12229.49 10163.71 22596.21 20203.90 17646.40 19760.48 19763.96
[289] 19472.24 31050.31 23749.96 30622.82 12132.51 NA 12875.97 NA
[297] NA NA 12912.70 12417.50 NA NA NA NA
[305] 13782.80 14407.52 NA 13775.80 NA 21500.87 15629.06 NA
[313] NA 13830.21 NA NA 30897.98 NA NA 26658.78
[321] NA NA 13380.44 NA NA NA 12926.68 NA
[329] 0.00 0.00 17234.70 NA 13646.85 NA NA 13608.18
[337] NA 12985.74 NA NA 13567.33 NA NA 22866.67
[345] 15341.17 15151.77 15140.54 26348.12 14915.86 15125.86 15374.81 14144.65
[353] 13745.90 13776.05 NA 13769.22 13873.82 15107.52 31308.12 28424.08
[361] 15472.52 14514.95 NA 14683.33 NA 14846.92 NA 12991.96
[369] NA 32738.81 32208.01 NA 16237.65 NA 31624.02 NA
[377] 16119.08 NA 15079.46 NA NA 14650.77 NA NA
[385] 16088.35 14792.69 14610.90 15545.47 14855.49 14266.13 30998.13 14335.63
[393] 31017.98 31344.02 31116.08 29234.29 28630.08 27774.08 15154.44 28493.28
[401] 15544.37 NA NA 8490.81 NA 10890.16 NA 7189.80
[409] NA 14315.92 17251.97 NA NA NA列を絞る
filter()である条件に合っている列だけに絞る
チャレンジ
flow_cyt_dataから”sample_peak”と”tomato_peak”のデータを切り出して、それぞれがゼロより大きい列だけに絞って下さい
データの変換
mutate()は新しい行を作ることができる
# A tibble: 414 × 4
id_num tomato_peak sample_peak sample_size
<dbl> <dbl> <dbl> <dbl>
1 1 3975. 6848. 3.38
2 2 5452. 8089. 2.91
3 3 5496. 7835. 2.79
4 4 5050. 8093. 3.14
5 5 5896. 0 0
6 6 5042. 0 0
7 7 4896. 0 0
8 8 NA NA NA
9 9 NA NA NA
10 10 NA NA NA
# ℹ 404 more rowsデータの変換
mutate()である行のデータを変換することもできる
# A tibble: 414 × 4
id_num tomato_peak sample_peak sample_size
<dbl> <dbl> <dbl> <dbl>
1 1 3975. 6848. 3376.
2 2 5452. 8089. 2908.
3 3 5496. 7835. 2794.
4 4 5050. 8093. 3141.
5 5 5896. 0 0
6 6 5042. 0 0
7 7 4896. 0 0
8 8 NA NA NA
9 9 NA NA NA
10 10 NA NA NA
# ℹ 404 more rowsパイプについて
今までのやり方では、複数の処理をする際、それぞれのアウトプットに名前をつけていた:
flow_cyt_dataから”sample_peak”と”tomato_peak”のデータを切り出して、それぞれがゼロより大きい列だけに絞って下さい
でもこれは処理が多くなるとややこしくなってくる。
もっといい方法はある:パイプ
パイプについて
「パイプ」とは、左の関数のアウトプットを右の関数の
インプットに渡すもの
%>%(あるいは|>)と書く。「それから」として読む
パイプについて
- 先の作業はパイプを使うとこのようにできる
# A tibble: 199 × 2
sample_peak tomato_peak
<dbl> <dbl>
1 6848. 3975.
2 8089. 5452.
3 7835. 5496.
4 8093. 5050.
5 9752. 6176.
6 8488. 5402.
7 10504. 6294.
8 10225. 5039.
9 11934. 5669.
10 11558. 5415.
# ℹ 189 more rows- この方がスッキリするでしょう?
チャレンジ
- パイプを使うことによって以下の段階を含むパイプラインを組んでください:
- 生データを読み込む
- 必要な行だけに絞る
- 欠測データを排除する
- ゲノムサイズを計算する
休憩
Part II: ggplot2による図の作成
ggplot2について
Rには備え付けの関数、
plot()があるが、今回はtidyverseのggplot2パッケージを使うgg= “Grammar of Graphics”(画像の文法)- 「文法」が分かれば、(ほとんど)
どんな図でも作れる
- 「文法」が分かれば、(ほとんど)
ggplot2について
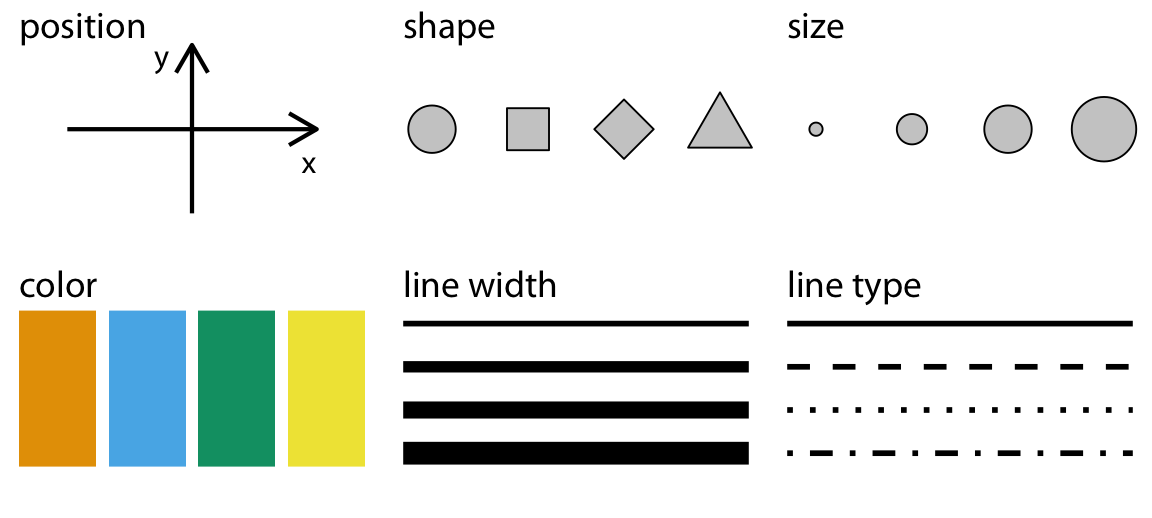
図の構造にはいくつか決まった要素がある
geometry: 図はどのような形にする?
aesthetics: データをどのように図に表す?
Geometry
Pie chart
Bar graph
Aesthetics
ggplot2の基本的な使い方

ggplot2の基本的な使い方

チャレンジ
sample_sizeを横軸、sample_peakを縦軸にして、点グラフを作成して下さい
倍数性の解析と可視化を一緒に
しよう
フローサイトメーターは直接に倍数性を教えてくれない。
このデータからどうやって倍数性を推測することができるのか?
まずはゲノムサイズの分布を可視化してみよう
ヒストグラムの作成
geom_histogram()でヒストグラムを作るaes()はxだけで良い
ヒストグラムの作成
- ビン(箱)の数はデフォルトで30
bins設定で変更可能
ヒストグラムの作成
geom_rug()を追加するとデータが実際にどこにあるのか、可視化する
チャレンジ
binsの値を色々試して見てください。どの設定が一番いいと思いますか?
倍数性の測定
- 倍数性とゲノムサイズは相関関係があるはず
- 4xは2xの倍の大きさ、など
- 先行研究では、イタドリは2x, 4x, 6x, 8xが知られている(とする)
チャレンジ
もう一度ヒストグラムを作成してください。
ヒストグラムのどこからどこまでがそれぞれの倍数性に一致すると思いますか?
休憩
Part III: kmeans法に
よる倍数性の分け方
チャレンジ
もう一度ヒストグラムを作成してください。
ヒストグラムのどこからどこまでがそれぞれの倍数性に一致すると思いますか?
グループ分けの仕方
目で見て判断するのはなかなか難しいですね。
適当に決めるのではなく、アルゴリズムを使ってみよう
グループ分けの仕方:kmeans
- kmeansとは、連続的なデータをグループ分けする
アルゴリズム
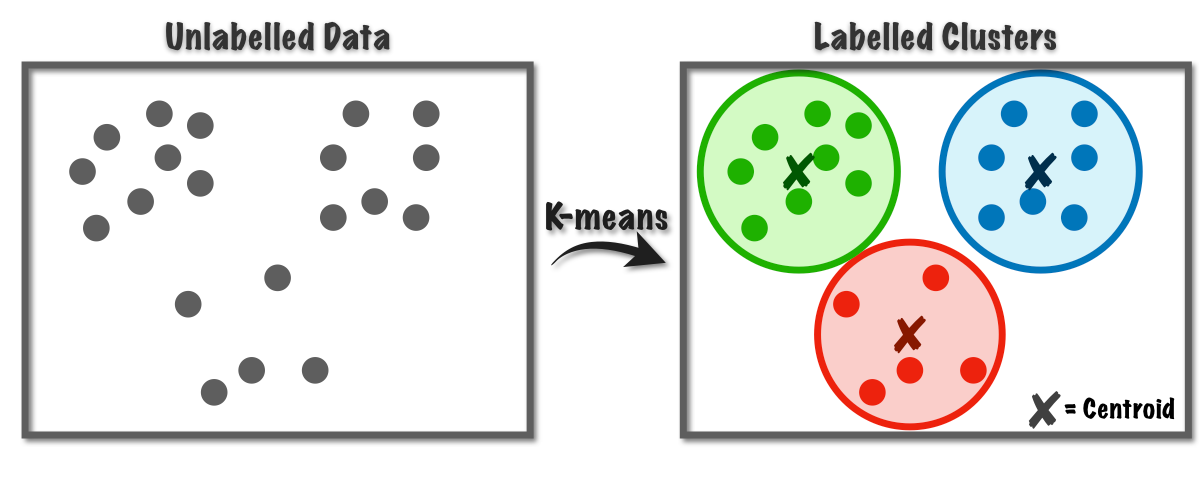
kmeansの仕組み
- 最初の重点をk個ランダムに置く(◯)
- 格データポイント(◾️)を最も近い重点(◯)によってグールプ分けする(色)
- それぞれのグループの新しい重点を計算する
- グループが変わらないまで 2.–3. を繰り返す

kmeansの仕組み

kmeansの仕組み:ポイント
- あらかじめグループの数(k)を設定する必要がある
- 始まりがランダム
- よって、毎回結果が違う
早速、倍数性データに使ってみよう
K-means clustering with 4 clusters of sizes 100, 31, 17, 51
Cluster means:
[,1]
1 4.031496
2 3.025890
3 6.073266
4 8.068650
Clustering vector:
[1] 2 2 2 2 2 2 2 1 1 1 1 4 1 2 2 2 1 4 4 3 2 2 1 1 1 1 3 1 2 2 1 1 1 1 1 1 1
[38] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 4 3 4 4 4 4 4 4 4 4 4 4 3 4 4 4 4 4
[75] 1 1 1 1 1 4 4 4 4 4 1 1 4 4 4 4 4 4 1 1 4 4 1 2 1 1 1 3 1 3 3 1 2 2 2 4 2
[112] 2 2 2 2 2 2 1 2 1 2 4 3 3 3 3 3 4 3 4 2 1 1 1 1 1 1 3 1 1 4 4 1 2 3 1 1 1
[149] 1 3 1 1 1 3 1 1 1 1 1 1 1 1 1 4 4 1 1 1 1 1 4 4 1 4 1 1 1 1 1 1 1 1 1 4 1
[186] 4 4 4 4 4 4 1 4 1 2 1 2 1 1
Within cluster sum of squares by cluster:
[1] 6.592609 2.527355 3.277437 6.267951
(between_SS / total_SS = 97.4 %)
Available components:
[1] "cluster" "centers" "totss" "withinss" "tot.withinss"
[6] "betweenss" "size" "iter" "ifault" チャレンジ
kmeans()関数をもう一度、全く同じように使ってみて、前の結果と比べてみてください。何かに気が付きましたか?
乱数生成
kmeans()はランダムなところからスタートすることによって、結果が毎回(ちょっと)違う- でも解析を再現したいときはどうすれば良い?毎回違うと困るのではないか?
- 解決方法:
set.seed()によって、ランダムな数字を固定させる
乱数生成
runif()は0-1の間のランダムな数字を返す
皆さんの結果はどうですか?私のと同じでしょうか?
乱数生成
set.seed()は、ランダムな数字を固定させる
今度はどうですか?
乱数生成
というわけで、kmeans()を使う前に、set.seed()を使いましょう。
kmeansによる倍数性のグループ分け
K-means clustering with 4 clusters of sizes 17, 51, 31, 100
Cluster means:
[,1]
1 6.073266
2 8.068650
3 3.025890
4 4.031496
Clustering vector:
[1] 3 3 3 3 3 3 3 4 4 4 4 2 4 3 3 3 4 2 2 1 3 3 4 4 4 4 1 4 3 3 4 4 4 4 4 4 4
[38] 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 2 1 2 2 2 2 2 2 2 2 2 2 1 2 2 2 2 2
[75] 4 4 4 4 4 2 2 2 2 2 4 4 2 2 2 2 2 2 4 4 2 2 4 3 4 4 4 1 4 1 1 4 3 3 3 2 3
[112] 3 3 3 3 3 3 4 3 4 3 2 1 1 1 1 1 2 1 2 3 4 4 4 4 4 4 1 4 4 2 2 4 3 1 4 4 4
[149] 4 1 4 4 4 1 4 4 4 4 4 4 4 4 4 2 2 4 4 4 4 4 2 2 4 2 4 4 4 4 4 4 4 4 4 2 4
[186] 2 2 2 2 2 2 4 2 4 3 4 3 4 4
Within cluster sum of squares by cluster:
[1] 3.277437 6.267951 2.527355 6.592609
(between_SS / total_SS = 97.4 %)
Available components:
[1] "cluster" "centers" "totss" "withinss" "tot.withinss"
[6] "betweenss" "size" "iter" "ifault" リストについて
kmeans()の出力はリストとなっている。
リストの要素を取り出すのに、$を使う:
図を作る時、このリストに入っているデータを使う。
分けたグループをプロットしよう
kmeansのグループを倍数性に当てる
分けたグループをプロットしよう
まずは、ポイント(点)だけ
でも点が重なっていて見づらい・・
- geom_point()ではなく、geom_jitter()を使いましょう
- geom_jitter()はポイントの位置を適当にバラす
わかりやすくなりました。
次に、またヒストグラムに戻りましょう。
geom_vline()(縦線を書く関数)を使って、重点の位置をプロットしよう
geom_rug()(マージンにデータをプロットする関数)を使って、ポイントの位置を確かめよう

- 倍数性プロットの完成!
チャレンジ
倍数性はこれでいいと思いますか?
怪しい点はありませんか?
まとめ
データとコードがあれば、計算結果の再現ができる
倍数性は目で適当に決めるよりも、アルゴリズムを使った方が客観的で再現性が高い
- でも、だからと言って絶対に正解だと言えない(そもそものデータにノイズが多すぎると、無理なこともある)
- 他のデータ(染色体観察など)で確かめる必要がある

Motsa, M.M., Bester, C., Slabbert, M.M. et al. Flow cytometry: a quick method to determine ploidy levels in honeybush (Cyclopia spp.). Genet Resour Crop Evol 65, 1711–1724 (2018).










