Content from RとRStudio入門
Last updated on 2025-07-01 | Edit this page
Estimated time: 55 minutes
Overview
Questions
- RStudio内でどのように操作するのか?
- Rとの対話方法は?
- 環境をどのように管理するのか?
- パッケージをどのようにインストールするのか?
Objectives
- RStudioの各ペインの目的と使用方法を説明する
- RStudio内のボタンやオプションの場所を見つける
- 変数を定義する
- データを変数に代入する
- インタラクティブなRセッションでワークスペースを管理する
- 数学および比較演算子を使用する
- 関数を呼び出す
- パッケージを管理する
ワークショップを始める前に
ワークショップで使用する一部のパッケージが正しくインストールされない(または全くインストールされない)場合があるため、お使いのマシンにRとRStudioの最新バージョンがインストールされていることを確認してください。
なぜRとRStudioを使うのか?
Software CarpentryワークショップのRセクションへようこそ!
科学は多段階のプロセスです。 実験を設計してデータを収集した後、本当の楽しみは分析から始まります!このレッスンでは、R言語の基本を教えるとともに、科学プロジェクトのためのコードを整理するベストプラクティスを学び、作業をより簡単にする方法を紹介します。
データを分析するためにMicrosoft ExcelやGoogleスプレッドシートを使用することもできますが、これらのツールは柔軟性やアクセス性に限界があります。さらに、元データの変更や探索の手順を共有することが難しいため、これは「再現可能な」研究にとって重要なポイントです(再現可能な研究についてはこちら)。
したがって、このレッスンでは、RとRStudioを使用してデータの探索を始める方法を学びます。RプログラムはWindows、Mac、Linuxオペレーティングシステムで利用可能で、上記のリンクから無料でダウンロードできます。Rを実行するために必要なのはRプログラムだけです。
しかし、Rをより使いやすくするために、同じくダウンロードしたRStudioというプログラムを使用します。RStudioは無料でオープンソースの統合開発環境(IDE)で、組み込みエディタを提供し、すべてのプラットフォームで動作します(サーバー上でも利用可能)。バージョン管理やプロジェクト管理との統合など、多くの利点があります。
概要
生データから始め、探索的な分析を行い、結果をグラフでプロットする方法を学びます。この例では、gapminder.orgのデータセットを使用し、時間を通じた各国の人口情報を扱います。データをRに読み込むことができますか?セネガルの人口をプロットできますか?アジア大陸の国々の平均所得を計算できますか?このレッスンの終わりまでに、これらの国々の人口を1分以内にプロットできるようになります!
基本レイアウト
RStudioを初めて開くと、次の3つのパネルが表示されます:
- インタラクティブなRコンソール/ターミナル(左全体)
- 環境/履歴/接続(右上のタブ)
- ファイル/プロット/パッケージ/ヘルプ/ビューア(右下のタブ)
ファイル(例えばRスクリプト)を開くと、上部左にエディタパネルも表示されます。
Rスクリプト
Rコンソールに書いたコマンドはファイルに保存し、再実行することができます。このようなRコードを含むファイルをRスクリプトと呼びます。Rスクリプトは名前の末尾が.Rとなっており、それがRスクリプトであることを示します。
RStudio内でのワークフロー
RStudio内で作業する主な方法は2つあります:
- インタラクティブなRコンソール内でテストや試行を行い、そのコードをコピーして.Rファイルに貼り付け、後で実行する。
- 小規模なテストや初期の段階では効果的です。
- しかし、すぐに手間がかかるようになります。
- 最初から.Rファイルに記述し、RStudioのショートカットキーを使用して「Run」コマンドを実行し、現在の行、選択した行、または変更した行をインタラクティブなRコンソールに送る。
- これは作業を始める良い方法です。すべてのコードが後で使用するために保存されます。
- RStudio内またはRの
source()関数を使用して作成したファイルを実行できます。
ヒント:コードのセグメントを実行する
RStudioでは、エディタウィンドウからコードを実行する柔軟性があります。ボタン、メニューオプション、およびキーボードショートカットがあります。現在の行を実行するには、次の方法があります:
- エディタパネルの上部にある「Run」ボタンをクリックする
- 「Code」メニューから「Run Lines」を選択する
- WindowsまたはLinuxではCtrl+Return、OS Xでは⌘+Returnを押す (このショートカットはボタンの上にマウスをホバーさせると確認できます)。コードブロックを実行するには、選択して「Run」をクリックします。 最近実行したコードブロックを修正した場合、セクションを再選択して「Run」を押す必要はありません。次のボタン「Re-run the previous region」を使用すると、修正を含む前のコードブロックを実行できます。
R入門
Rでの作業の多くは、インタラクティブなRコンソール内で行います。ここでは、すべてのコードを実行し、Rスクリプトファイルに追加する前にアイデアを試すのに便利な環境です。RStudioのコンソールは、コマンドライン環境でRと入力した場合と同じです。
Rのインタラクティブセッションを開くと、最初に情報が表示され、その後に「>」と点滅するカーソルが現れます。これは、シェルレッスンで学んだシェル環境と多くの点で似ています。「Read, evaluate, print loop」(読み取り、評価、印刷ループ)の考え方に基づいて動作します:コマンドを入力すると、Rがそれを実行し、結果を返します。
Rを計算機として使う
Rで最も簡単なことは、算術を行うことです:
R
1 + 100
OUTPUT
[1] 101Rは答えを表示し、その前に”[1]“を付けます。[1]はコンソールに表示される行の最初の要素のインデックスを示します。ベクトルのインデックスについての詳細は、エピソード6:データのサブセット化を参照してください。
不完全なコマンドを入力すると、R
は完了を待機します。Unix Shellのbashに慣れている場合、この動作をbashで見たことがあるかもしれません。
OUTPUT
+「>」ではなく「+」が表示された場合、Rはコマンドの完了を待機しています。コマンドをキャンセルしたい場合はEscを押すと、RStudioは「>」プロンプトに戻ります。
ヒント:コマンドのキャンセル
コマンドラインからRを使用している場合は、RStudioの代わりにEscではなくCtrl+Cを使用してコマンドをキャンセルする必要があります。これはMacユーザーにも適用されます!
コマンドのキャンセルは、不完全なコマンドを終了させるだけでなく、予想以上に時間がかかる場合にコードの実行を停止したり、現在書いているコードを削除したりするためにも役立ちます。
Rを計算機として使用する場合、演算の順序は学校で学んだものと同じです。
優先順位が高いものから低いものへ:
- 括弧:
(,) - 累乗:
^または** - 乗算:
* - 除算:
/ - 加算:
+ - 減算:
-
R
3 + 5 * 2
OUTPUT
[1] 13評価の順序を変更したい場合や意図を明確にしたい場合は、括弧を使用してグループ化します。
R
(3 + 5) * 2
OUTPUT
[1] 16必要ない場合は煩雑になりますが、意図を明確にできます。他の人が後でコードを読むかもしれないことを忘れないでください。
R
(3 + (5 * (2 ^ 2))) # 読みにくい
3 + 5 * 2 ^ 2 # 規則を覚えていれば明快
3 + 5 * (2 ^ 2) # 規則を忘れた場合はこれが助けになる
各コード行の後にあるテキストは「コメント」と呼ばれます。ハッシュ記号#の後に続く内容は、コードを実行する際にRによって無視されます。
非常に小さいまたは大きい数値は、科学表記法で表示されます:
R
2/10000
OUTPUT
[1] 2e-04これは「10^XXで掛ける」という短縮形です。したがって、2e-4は2 * 10^(-4)の短縮形です。
科学表記法で数値を書くこともできます:
R
5e3 # マイナスがない点に注意
OUTPUT
[1] 5000数学関数
Rには多くの組み込み数学関数があります。関数を呼び出すには、関数名を入力し、その後に開き括弧と閉じ括弧を続けます。関数は引数を入力として受け取ります。関数の括弧内に入力したものはすべて引数と見なされます。関数によって引数の数は異なり、引数を必要としないものから複数の引数を必要とするものまであります。例:
R
getwd() # 絶対パスを返す
この例では引数は不要ですが、以下の数学関数では結果を計算するために値を渡す必要があります。
R
sin(1) # 三角関数
OUTPUT
[1] 0.841471R
log(1) # 自然対数
OUTPUT
[1] 0R
log10(10) # 常用対数(底10)
OUTPUT
[1] 1R
exp(0.5) # e^(1/2)
OUTPUT
[1] 1.648721Rのすべての関数を覚えようとする必要はありません。Googleで検索するか、関数名の最初の数文字を覚えていれば、RStudioのタブ補完機能を使うことができます。
RStudioの大きな利点の一つは、オートコンプリート機能があることです。これにより、関数、引数、および受け取る値を簡単に調べることができます。
コマンド名の前に?を付けると、そのコマンドのヘルプページが開きます。RStudioを使用している場合、’Help’ペインに表示されます。ターミナルでRを使用している場合は、ブラウザでヘルプページが開きます。ヘルプページにはコマンドの詳細な説明と動作の仕組みが含まれています。ページの下部までスクロールすると、通常、コマンドの使用例が掲載されています。後ほど例を見ていきます。
比較演算
Rでは比較を行うこともできます:
R
1 == 1 # 等しい(等号が2つ、"等しい"と読む)
OUTPUT
[1] TRUER
1 != 2 # 等しくない("等しくない"と読む)
OUTPUT
[1] TRUER
1 < 2 # より小さい
OUTPUT
[1] TRUER
1 <= 1 # 以下
OUTPUT
[1] TRUER
1 > 0 # より大きい
OUTPUT
[1] TRUER
1 >= -9 # 以上
OUTPUT
[1] TRUEヒント:数値の比較
数値を比較する際の注意点として、整数(小数を含まない数値型)以外を比較する場合は、==を使用しないでください。
コンピュータは小数を特定の精度でしか表現できないため、Rが表示する際に同じに見える2つの数値が、内部表現では異なる場合があります。このわずかな差異は「数値計算誤差(Machine numeric tolerance)」と呼ばれます。
代わりにall.equal関数を使用してください。
さらに詳しく知りたい方はこちら:http://floating-point-gui.de/
変数と代入
代入演算子<-を使用して、値を変数に格納できます:
R
x <- 1/40
代入は値を表示しません。代わりに、それを後で使用するために変数というものに格納します。この場合、xには値0.025が格納されています:
R
x
OUTPUT
[1] 0.025正確には、この格納された値は浮動小数点数と呼ばれる分数の10進数近似値です。
RStudioの右上ペインにあるEnvironmentタブを確認すると、xとその値が表示されていることがわかります。変数xは、数値を期待する計算の中で数値の代わりに使用できます:
R
log(x)
OUTPUT
[1] -3.688879また、変数には再代入も可能です:
R
x <- 100
以前はxに0.025が格納されていましたが、現在は100が格納されています。
代入値には、代入先の変数を含めることもできます:
R
x <- x + 1 # RStudioの右上タブでxの説明が更新されることに注目
y <- x * 2
代入の右辺には有効なR式を使用できます。右辺は代入が行われる前に完全に評価されます。
変数名には、文字、数字、アンダースコア、ピリオドを含めることができますが、スペースは含められません。また、変数名は文字またはピリオドで始める必要があります(数字やアンダースコアでは始めることはできません)。ピリオドで始まる変数は隠し変数と見なされます。
長い変数名については、異なる人が異なる規約を使用します。その例として:
- periods.between.words
- underscores_between_words
- camelCaseToSeparateWords
どれを使用するかは自由ですが、一貫性を保つことが重要です。
代入には=演算子を使用することも可能です:
R
x = 1/40
しかし、これはRユーザーの間ではあまり一般的ではありません。最も重要なのは、使用する演算子に一貫性を持つことです。<-を使用したほうが混乱が少ない場合もあり、コミュニティでは最も一般的に使われています。そのため、<-を使用することを推奨します。
ベクトル化
Rの特徴の1つに、Rがベクトル化されているという点があります。つまり、変数や関数にベクトルを値として持たせることができます。物理学や数学におけるベクトルとは異なり、Rにおけるベクトルは同じデータ型の値が順序付けられた集合を指します。例:
R
1:5
OUTPUT
[1] 1 2 3 4 5R
2^(1:5)
OUTPUT
[1] 2 4 8 16 32R
x <- 1:5
2^x
OUTPUT
[1] 2 4 8 16 32この機能は非常に強力で、今後のレッスンでさらに詳しく説明します。
環境の管理
Rセッションとやり取りするための便利なコマンドがいくつかあります。
lsを使用すると、グローバル環境(現在のRセッション)に保存されているすべての変数と関数を一覧表示できます:
R
ls()
OUTPUT
[1] "x" "y"ヒント:隠しオブジェクト
シェルと同様に、lsではデフォルトで”.”で始まる変数や関数は表示されません。すべてのオブジェクトを一覧表示するには、ls(all.names=TRUE)と入力してください。
ここでは、lsに引数を渡していませんが、関数を呼び出すために括弧は必要です。
lsだけを入力すると、オブジェクト一覧ではなくコードが表示されます:
R
ls
OUTPUT
function (name, pos = -1L, envir = as.environment(pos), all.names = FALSE,
pattern, sorted = TRUE)
{
if (!missing(name)) {
pos <- tryCatch(name, error = function(e) e)
if (inherits(pos, "error")) {
name <- substitute(name)
if (!is.character(name))
name <- deparse(name)
warning(gettextf("%s converted to character string",
sQuote(name)), domain = NA)
pos <- name
}
}
all.names <- .Internal(ls(envir, all.names, sorted))
if (!missing(pattern)) {
if ((ll <- length(grep("[", pattern, fixed = TRUE))) &&
ll != length(grep("]", pattern, fixed = TRUE))) {
if (pattern == "[") {
pattern <- "\\["
warning("replaced regular expression pattern '[' by '\\\\['")
}
else if (length(grep("[^\\\\]\\[<-", pattern))) {
pattern <- sub("\\[<-", "\\\\\\[<-", pattern)
warning("replaced '[<-' by '\\\\[<-' in regular expression pattern")
}
}
grep(pattern, all.names, value = TRUE)
}
else all.names
}
<bytecode: 0x555f519a3fc0>
<environment: namespace:base>これはどういうことでしょうか?
Rではすべてがオブジェクトであり、オブジェクト名だけを入力すると、その内容が表示されます。先ほど作成したオブジェクトxには1,
2, 3, 4, 5が格納されています:
R
x
OUTPUT
[1] 1 2 3 4 5オブジェクトlsには、ls関数を動作させるRコードが格納されています!関数の仕組みや作成方法については後のレッスンで説明します。
不要になったオブジェクトを削除するには、rmを使用します:
R
rm(x)
多くのオブジェクトが環境にあり、それらをすべて削除したい場合は、lsの結果をrm関数に渡します:
R
rm(list = ls())
この場合、2つの関数を組み合わせています。演算の順序と同様に、最も内側の括弧内の内容が最初に評価されます。
この場合、lsの結果がrmのlist引数として使用されるよう指定しています。引数に値を名前で割り当てる場合、必ず=演算子を使用する必要があります!
代わりに<-を使用すると、予期しない副作用が発生するか、エラーメッセージが表示される可能性があります:
R
rm(list <- ls())
ERROR
Error in rm(list <- ls()): ... must contain names or character stringsヒント:警告とエラー
Rが予期しない動作をした場合は注意してください!エラーはRが計算を続行できない場合に発生します。一方、警告は通常、関数が実行されたものの、期待通りに動作しなかったことを意味します。
どちらの場合も、Rが表示するメッセージには問題を解決するための手がかりが含まれていることが多いです。
Rパッケージ
Rにはパッケージを作成することで関数を追加することができます。また、他の人が作成したパッケージを利用することも可能です。この執筆時点で、CRAN(Comprehensive R Archive Network)には10,000を超えるパッケージが利用可能です。RとRStudioにはパッケージを管理するための機能があります:
- インストールされているパッケージを確認するには、
installed.packages()を入力します。 - パッケージをインストールするには、
install.packages("packagename")と入力します。ここでpackagenameはパッケージ名で、引用符で囲みます。 - インストール済みのパッケージを更新するには、
update.packages()を入力します。 - パッケージを削除するには、
remove.packages("packagename")を使用します。 - パッケージを利用可能にするには、
library(packagename)を入力します。
RStudioの右下ペインの「Packages」タブからもパッケージを表示、ロード、デタッチすることができます。このタブをクリックすると、インストール済みのパッケージがチェックボックス付きで表示されます。パッケージ名の横にあるチェックボックスがオンの場合、そのパッケージはロードされており、オフの場合はロードされていません。空のボックスをクリックするとそのパッケージがロードされ、チェックボックスをクリックするとパッケージがデタッチされます。
また、「Packages」タブの上部にある「Install」ボタンと「Update」ボタンを使用して、パッケージをインストールおよび更新できます。
チャレンジ 2
次のプログラムの各文の後で、各変数の値はどうなるでしょうか?
R
mass <- 47.5
age <- 122
mass <- mass * 2.3
age <- age - 20
R
mass <- 47.5
この時点で変数massの値は47.5になります。
R
age <- 122
この時点で変数ageの値は122になります。
R
mass <- mass * 2.3
既存の値47.5に2.3を掛け、新しい値109.25をmassに格納します。
R
age <- age - 20
既存の値122から20を引き、新しい値102をageに格納します。
チャレンジ 3
前のチャレンジのコードを実行し、massとageを比較するコマンドを書きなさい。massはageより大きいですか?
この質問に答える方法の1つとして、次のように>を使用できます:
R
mass > age
OUTPUT
[1] TRUEこのコードは、109.25が102より大きいため、論理値TRUEを返すはずです。
チャレンジ 4
作業環境を整理し、massとageの変数を削除しなさい。
このタスクを達成するには、rmコマンドを使用します:
R
rm(age, mass)
チャレンジ 5
以下のパッケージをインストールしなさい:ggplot2,
plyr, gapminder
必要なパッケージをインストールするには、install.packages()コマンドを使用します。
R
install.packages("ggplot2")
install.packages("plyr")
install.packages("gapminder")
1つのinstall.packages()コマンドで複数のパッケージ
をインストールする別の方法は次のとおりです:
R
install.packages(c("ggplot2", "plyr", "gapminder"))
ggplot2をインストールする際、一部のユーザーは依存関係フラグを使用する必要がある場合があります。これは既知のバグではなく、ワークショップの実施中に確認されたエラーを解決するための推奨事項です:
R
install.packages("ggplot2", dependencies = TRUE)
- RStudioを使用してRプログラムを作成および実行します。
- Rには通常の算術演算子と数学関数があります。
-
<-を使用して変数に値を代入します。 -
ls()を使用してプログラム内の変数を一覧表示します。 -
rm()を使用してプログラム内のオブジェクトを削除します。 -
install.packages()を使用してパッケージ(ライブラリ)をインストールします。
Content from RStudio を使ったプロジェクト管理
Last updated on 2025-07-01 | Edit this page
Estimated time: 30 minutes
Overview
Questions
- R でプロジェクトをどのように管理できますか?
Objectives
- RStudio で自己完結型のプロジェクトを作成する
はじめに
科学的なプロセスは本質的に段階的なものであり、多くのプロジェクトはランダムなメモ、一部のコード、次に原稿と進行し、最終的にはすべてが混ざり合ってしまうことがよくあります。
プロジェクトを再現可能な方法で管理することは、科学を再現可能にするだけでなく、生活をより簡単にします。
— Vince Buffalo (@vsbuffalo) 2013年4月15日
ほとんどの人はプロジェクトを次のように整理しがちです:
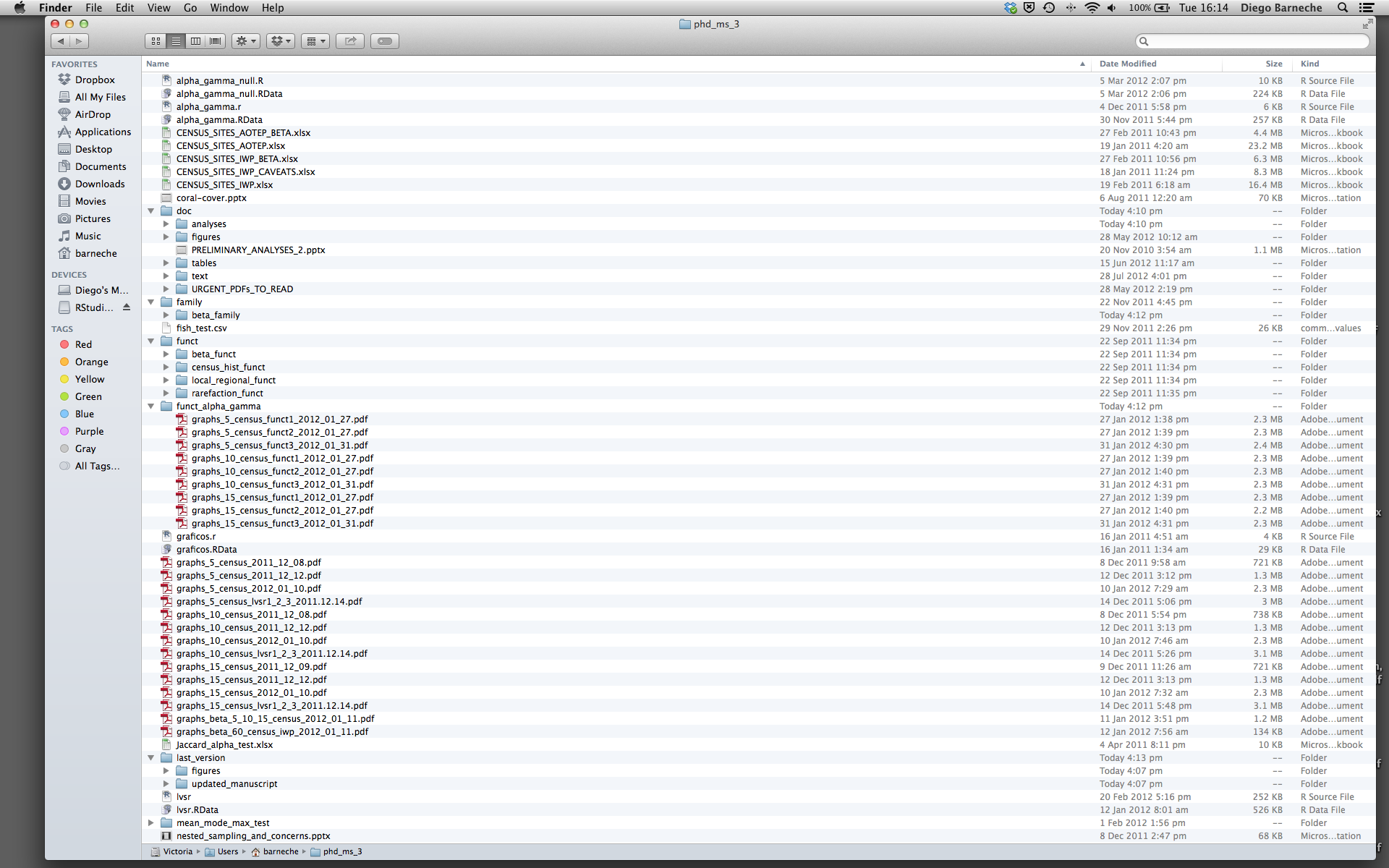
このような方法を絶対に避けるべき理由は数多くあります:
- データのどのバージョンがオリジナルで、どれが修正済みなのかを区別するのが非常に難しい。
- 様々な拡張子のファイルが混在して、非常に散らかる。
- 必要なものを見つけたり、正確なコードで生成した正しい図表を関連付けたりするのに非常に時間がかかる。
良いプロジェクト構成は、最終的に生活をより簡単にします:
- データの整合性を確保しやすくなる。
- 他の人(研究室の同僚、共同研究者、指導教員)とコードを共有するのが簡単になる。
- 原稿の投稿時にコードを簡単にアップロードできる。
- しばらく休んだ後にプロジェクトを再開しやすくなる。
考えられる解決策
幸いなことに、作業を効果的に管理するためのツールやパッケージが存在します。
RStudio の最も強力で便利な機能の一つがプロジェクト管理機能です。本日はこれを使って自己完結型の再現可能なプロジェクトを作成します。
チャレンジ 1: 自己完結型プロジェクトの作成
RStudio で新しいプロジェクトを作成します:
- 「File」メニューをクリックし、「New Project」を選択します。
- 「New Directory」をクリックします。
- 「New Project」をクリックします。
- プロジェクトを保存するディレクトリの名前(例:
my_project)を入力します。 - 「Create a git repository」のチェックボックスが表示される場合は選択します。
- 「Create Project」ボタンをクリックします。
作成した RStudio
プロジェクトを開く最も簡単な方法は、ファイルシステムをたどって保存したディレクトリに移動し、.Rproj
ファイルをダブルクリックすることです。これにより RStudio が開き、R
セッションが .Rproj
ファイルと同じディレクトリで開始します。データ、プロット、スクリプトはすべてプロジェクトディレクトリに関連付けられます。さらに、RStudio
プロジェクトは複数のプロジェクトを同時に開くことが可能で、それぞれのプロジェクトディレクトリに分離されます。これにより、複数のプロジェクトを開いても相互に干渉しません。
チャレンジ 2: ファイルシステムを使った RStudio プロジェクトの開き方
- RStudio を終了します。
- チャレンジ 1 で作成したプロジェクトのディレクトリに移動します。
- そのディレクトリ内の
.Rprojファイルをダブルクリックします。
プロジェクト管理のベストプラクティス
プロジェクトを整理するための「ベスト」な方法はありませんが、管理を容易にするために従うべきいくつかの一般原則があります:
データを読み取り専用として扱う
プロジェクトを設定する際の最も重要な目標はこれです。データの収集は通常、時間と費用がかかります。データをインタラクティブに操作する(例:Excel で)と、データの出所や収集後にどのように変更されたかを把握できなくなります。そのため、データを「読み取り専用」として扱うのが良い考えです。
データのクリーニング
多くの場合、データは「汚れて」おり、R(または他のプログラミング言語)で有用な形式にするために大幅な前処理が必要です。このタスクは「データマンジング」と呼ばれることもあります。これらのスクリプトを別のフォルダに保存し、クリーンなデータセットを保持する「読み取り専用」データフォルダを作成することで、両者の混同を防ぐことができます。
生成された出力を使い捨てとみなす
スクリプトによって生成されたものはすべて使い捨てとみなすべきです:スクリプトからすべてを再生成できる必要があります。
出力を管理する方法はたくさんあります。各分析ごとに異なるサブディレクトリを持つ出力フォルダを用意すると、後で便利です。多くの分析は探索的で最終プロジェクトに使用されないことが多く、一部の分析はプロジェクト間で共有されることもあります。
ヒント: 科学的コンピューティングのための「十分に良い」実践
科学的コンピューティングのための「十分に良い」実践 では、プロジェクトの構成について以下の推奨事項を挙げています:
- 各プロジェクトを専用のディレクトリに配置し、そのディレクトリにプロジェクト名を付ける。
- プロジェクトに関連するテキスト文書を
docディレクトリに配置する。 - 生データとメタデータを
dataディレクトリに、クリーンアップや分析中に生成されたファイルをresultsディレクトリに配置する。 - プロジェクトのスクリプトやプログラムのソースを
srcディレクトリに配置し、他から持ち込んだプログラムやローカルでコンパイルしたプログラムをbinディレクトリに配置する。 - すべてのファイルに内容や機能を反映した名前を付ける。
関数の定義と適用を分離する
R を効率的に使用する最も効果的な方法の一つは、最初に .R スクリプトに実行したいコードを書き、RStudio のキーボードショートカットを使用するか「Run」ボタンをクリックして、選択した行をインタラクティブな R コンソールで実行することです。
プロジェクトの初期段階では、最初の .R スクリプトファイルに多くの直接実行されるコード行が含まれることがよくあります。プロジェクトが進むにつれて、再利用可能なコードチャンクが独自の関数に分離されます。これらの関数を保存するためのフォルダと分析スクリプトを保存するためのフォルダを分けるのが良いアイデアです。
データを data ディレクトリに保存する
良好なディレクト
リ構造ができたら、データファイルを data/
ディレクトリに保存します。
チャレンジ 3
このリンクから CSV ファイルをダウンロードしてください。
- ファイルをダウンロードします(上記リンクを右クリック -> 「リンク先を名前を付けて保存」/「名前を付けて保存」、またはリンクをクリックしページが読み込まれた後に Ctrl+S を押すか、メニューの「ファイル」 -> 「ページを名前を付けて保存」を選択)。
-
gapminder_data.csvという名前で保存されていることを確認します。 - ファイルをプロジェクト内の
data/フォルダに保存します。
後ほどこのデータを読み込み、確認します。
チャレンジ 4
R に読み込む前に、コマンドラインからデータセットについての一般的な情報を得ることは有益です。これにより、R に読み込む際の判断に役立ちます。コマンドラインシェルを使用して以下の質問に答えてください:
- ファイルのサイズはどれくらいですか?
- このファイルには何行のデータがありますか?
- このファイルにはどのような値が含まれていますか?
次のコマンドをシェルで実行します:
OUTPUT
-rw-r--r-- 1 runner docker 80K Jul 1 00:26 data/gapminder_data.csvファイルサイズは 80K です。
OUTPUT
1705 data/gapminder_data.csv行数は 1705 行です。データの内容は次のようになります:
OUTPUT
country,year,pop,continent,lifeExp,gdpPercap
Afghanistan,1952,8425333,Asia,28.801,779.4453145
Afghanistan,1957,9240934,Asia,30.332,820.8530296
Afghanistan,1962,10267083,Asia,31.997,853.10071
Afghanistan,1967,11537966,Asia,34.02,836.1971382
Afghanistan,1972,13079460,Asia,36.088,739.9811058
Afghanistan,1977,14880372,Asia,38.438,786.11336
Afghanistan,1982,12881816,Asia,39.854,978.0114388
Afghanistan,1987,13867957,Asia,40.822,852.3959448
Afghanistan,1992,16317921,Asia,41.674,649.3413952ヒント: RStudio のコマンドライン
RStudio のコンソールペインにある「Terminal」タブを使用すると、RStudio 内で直接コマンドラインを操作できます。
作業ディレクトリ
R の現在の作業ディレクトリを知ることは重要です。なぜなら、他のファイルにアクセスする必要があるとき(例:データファイルをインポートする場合)、R は現在の作業ディレクトリを基準にそれらのファイルを探すからです。
新しい RStudio
プロジェクトを作成するたびに、そのプロジェクトの新しいディレクトリが作成されます。既存の
.Rproj ファイルを開くと、そのプロジェクトが開き、R
の作業ディレクトリがそのファイルがあるフォルダに設定されます。
チャレンジ 5
getwd() コマンドを使用するか、RStudio
のメニューを使って現在の作業ディレクトリを確認します。
- コンソールで
getwd()(“wd” は “working directory” の略)と入力し、Enter を押します。 - ファイルペインで、
dataフォルダをダブルクリックして開く(または他の任意のフォルダに移動)。作業ディレクトリに戻るには、ファイルペインの「More」をクリックし、「Go To Working Directory」を選択します。
setwd() コマンドを使用するか、RStudio
のメニューを使って作業ディレクトリを変更します。
- コンソールで
setwd("data")と入力し、Enter を押します。その後、getwd()と入力して Enter を押し、新しい作業ディレクトリを確認します。 - RStudio ウィンドウ上部のメニューで「Session」をクリックし、「Set
Working Directory」を選択して「Choose
Directory」をクリックします。その後、開いたウィンドウでプロジェクトディレクトリに戻り、「Open」をクリックします。コンソールに
setwdコマンドが自動的に表示されます。
ヒント: ファイルが存在しないエラー
R コードでファイルを参照しようとして「ファイルが存在しない」というエラーが出た場合は、作業ディレクトリを確認するのが良いです。 ファイルへの絶対パスを指定するか、作業ディレクトリ内(またはそのサブフォルダ)にファイルを保存し、相対パスを指定する必要があります。
バージョン管理
プロジェクトではバージョン管理を使用することが重要です。RStudio で Git を使用する方法についての良いレッスンはこちらを参照してください。
- RStudio を使用して一貫したレイアウトでプロジェクトを作成および管理する。
- 生データを読み取り専用として扱う。
- 生成された出力を使い捨てとみなす。
- 関数の定義と適用を分離する。
Content from ヘルプの利用
Last updated on 2025-07-01 | Edit this page
Estimated time: 20 minutes
Overview
Questions
- R でどのようにしてヘルプを得ることができますか?
Objectives
- 関数や特殊な演算子に関する R のヘルプファイルを読むことができる。
- 問題を解決するためのパッケージを特定するために CRAN タスクビューを利用できる。
- 仲間に助けを求める方法を理解する。
ヘルプファイルを読む
R および各パッケージには関数用のヘルプファイルが用意されています。特定のパッケージに含まれる関数についてヘルプを検索する際の一般的な構文は次の通りです:
R
?function_name
help(function_name)
たとえば、write.table()
のヘルプファイルを見てみましょう。この関数に似た機能を持つ関数を今後のエピソードで使用します。
R
?write.table()
これにより、RStudio ではヘルプページが表示され、R 本体ではプレーンテキストとして表示されます。
各ヘルプページは次のようなセクションに分かれています:
- Description(説明): 関数が何をするかの詳細な説明。
- Usage(使用法): 関数の引数とそのデフォルト値(変更可能)。
- Arguments(引数): 各引数が期待するデータの説明。
- Details(詳細): 注意すべき重要な点。
- Value(戻り値): 関数が返すデータ。
- See Also(関連項目): 有用な関連関数。
- Examples(例): 関数の使用例。
関数によってはセクションが異なる場合がありますが、これらが主なポイントです。
関連する関数が同じヘルプファイルを参照する場合があることに注意してください:
R
?write.table()
?write.csv()
これらの関数は非常に似た用途を持ち、引数も共通しているため、パッケージ作者が同じヘルプファイルで文書化していることがよくあります。
ヒント: 実例を実行する
ヘルプページ内の Examples セクションからコードをハイライトして Ctrl+Return を押すと、RStudio コンソールで実行されます。 関数の動作を素早く理解する方法です。
ヒント: ヘルプファイルを読む
R の大きな課題の一つは、利用可能な関数の数が膨大であることです。 すべての関数の正しい使用法を記憶するのは現実的ではありません。 しかし、ヘルプファイルを利用すれば、記憶する必要はありません!
特殊な演算子
特殊な演算子に関するヘルプを検索するには、引用符またはバッククォートを使用します:
R
?"<-"
?`<-`
パッケージに関するヘルプ
多くのパッケージには「ビネット」と呼ばれるチュートリアルや拡張的な例のドキュメントが含まれています。
引数なしで vignette()
を実行すると、インストール済みのすべてのパッケージのビネットが表示されます。
特定のパッケージについては vignette(package="パッケージ名")
を使用します。 特定のビネットを開くには
vignette("ビネット名") を実行します。
パッケージにビネットがない場合は、通常、次のコマンドでヘルプを探せます:
R
help("パッケージ名")
また、RStudio には多くのパッケージ向けに優れた チートシート があります。
関数名の一部を覚えている場合
関数がどのパッケージに属しているか、または正確なスペルがわからない場合、ファジー検索が可能です:
R
??function_name
ファジー検索では文字列の近似一致を検索します。たとえば、作業ディレクトリを設定する関数に「set」が含まれていることを覚えている場合、次のように検索できます:
R
??set
どこから始めるべきかわからない場合
どの関数やパッケージを使用すべきかわからない場合、 CRAN Task Views を利用するとよいでしょう。 これは、パッケージを分野別にグループ化した特別なリストで、出発点として適しています。
コードが動作しない場合: 仲間に助けを求める
関数の使用に問題がある場合、その答えのほとんどはすでに Stack Overflow
で回答されています。 [r]
タグを使って検索してください。質問の仕方については、Stack Overflow の 良い質問の仕方のページを参照してください。
答えが見つからない場合、以下の便利な関数を使って仲間に助けを求めるとよいでしょう:
R
?dput
この関数は、使用しているデータを他の人が自分の R セッションでコピー&ペーストできる形式に出力します。
R
sessionInfo()
OUTPUT
R version 4.5.1 (2025-06-13)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.5 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.10.0
LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.10.0 LAPACK version 3.10.0
locale:
[1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
[4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
[7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
[10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
time zone: UTC
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] compiler_4.5.1 tools_4.5.1 yaml_2.3.10 knitr_1.48 xfun_0.49
[6] renv_1.1.4 evaluate_1.0.1この関数は、R の現在のバージョンやロードしているパッケージを表示します。 これは他の人が問題を再現し、デバッグするのに役立ちます。
チャレンジ 1
c
関数のヘルプページを見てください。次のコードを評価した場合、どのようなベクトルが作成されると思いますか?
R
c(1, 2, 3)
c('d', 'e', 'f')
c(1, 2, 'f')
c()
関数はすべての要素が同じ型のベクトルを作成します。最初の場合、要素は数値型、
2 番目の場合は文字型、そして 3
番目の場合も文字型です。数値型の値は文字型に「強制変換」されます。
チャレンジ 2
paste
関数のヘルプを見てください。この関数を後ほど使用します。
sep 引数と collapse 引数の違いは何ですか?
paste() 関数のヘルプを見るには以下を実行します:
R
help("paste")
?paste
sep と collapse
の違いは少し複雑です。paste
関数は任意の数の引数を受け取り、それぞれが任意の長さのベクトルであることができます。
sep
引数は連結される各項目の間に使用される文字列を指定します(デフォルトはスペース)。
結果は、paste
に渡された最も長い引数と同じ長さのベクトルです。
一方、collapse
引数は、連結後の要素を指定された区切り文字を使用して「まとめて結合」することを示します。
その結果、単一の文字列になります。
引数を明示的に指定することが重要です。たとえば sep = ","
と入力すると、関数は区切り文字として “,”
を使用し、結合する項目としてではないと認識します。
例:
R
paste(c("a","b"), "c")
OUTPUT
[1] "a c" "b c"R
paste(c("a","b"), "c", ",")
OUTPUT
[1] "a c ," "b c ,"R
paste(c("a","b"), "c", sep = ",")
OUTPUT
[1] "a,c" "b,c"R
paste(c("a","b"), "c", collapse = "|")
OUTPUT
[1] "a c|b c"R
paste(c("a","b"), "c", sep = ",", collapse = "|")
OUTPUT
[1] "a,c|b,c"(詳細については、?paste
ヘルプページの末尾の例を参照するか、example('paste')
を試してください。)
チャレンジ 3
ヘルプを使用して、タブ区切り(\t)の列と小数点が
“.”(ピリオド)で表される表形式のファイルを読み込むために使用できる関数(および関連するパラメータ)を見つけてください。
特に国際的な同僚と協力している場合、小数点の表記(例:コンマ対ピリオド)は異なる場合があるため、この確認が重要です。
ヒント:??"read table"
を使用して表形式データの読み込みに関連する関数を調べてください。
タブ区切りファイルを小数点がピリオドで表される形式で読み込む標準的な
R 関数は read.delim() です。
また、read.table(file, sep="\t")
を使用することもできます(read.table()
のデフォルトの小数点はピリオドです)。
ただし、データファイルにハッシュ(#)文字が含まれている場合は、comment.char
引数を変更する必要があるかもしれません。
その他のリソース
- R のオンラインヘルプを取得するには
help()を使用します。
Content from データ構造
Last updated on 2025-07-01 | Edit this page
Estimated time: 55 minutes
Overview
Questions
- R でデータをどのように読み取ることができますか?
- R の基本的なデータ型は何ですか?
- R でカテゴリ情報をどのように表現しますか?
Objectives
- 5 つの主なデータ型を特定できるようになる。
- データフレームを探索し始め、ベクトルやリストとの関連を理解する。
- R からオブジェクトの型、クラス、構造に関する質問ができるようになる。
- “names”、“class”、“dim” 属性の情報を理解する。
R の最も強力な機能の 1 つは、スプレッドシートや CSV
ファイルにすでに保存されているような表形式データを処理する能力です。まずは、data/
ディレクトリに feline-data.csv
という名前の小さなデータセットを作成しましょう:
R
cats <- data.frame(coat = c("calico", "black", "tabby"),
weight = c(2.1, 5.0, 3.2),
likes_catnip = c(1, 0, 1))
次に、cats を CSV
ファイルとして保存します。引数名を明示的に指定することは良い習慣であり、関数が変更されたデフォルト値を認識できます。この場合は
row.names = FALSE
を設定しています。引数名やそのデフォルト値を確認するには、?write.csv
を使用してヘルプファイルを表示してください。
R
write.csv(x = cats, file = "data/feline-data.csv", row.names = FALSE)
新しいファイル feline-data.csv
の内容は次の通りです:
ヒント: R でテキストファイルを編集する
または、テキストエディタ(Nano)や RStudio の File -> New
File -> Text File メニュー項目を使用して
data/feline-data.csv を作成することもできます。
このデータを R に読み込むには、以下のコマンドを使用します:
R
cats <- read.csv(file = "data/feline-data.csv")
cats
OUTPUT
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1read.table 関数は、CSV ファイル(csv = comma-separated
values)などのテキストファイルに保存された表形式データを読み取るために使用されます。タブやカンマは、CSV
ファイルでデータポイントを区切るために最も一般的に使用される記号です。R
では read.table の便利なバージョンとして
read.csv(データがカンマで区切られている場合)と
read.delim(データがタブで区切られている場合)が用意されています。この
3 つの中で、read.csv
が最も一般的に使用されます。必要に応じて、デフォルトの区切り記号を変更することもできます。
データが因子かどうかを確認する
最近、R がテキストデータを処理する方法が変更されました。以前は、R はテキストデータを自動的に “因子” という形式に変換していましたが、現在は “文字列” という形式で処理されるようになりました。因子の使用用途については後ほど学びますが、ほとんどの場合は必要なく、使用することで複雑になるだけです。そのため、新しい R バージョンではテキストデータが “文字列” として読み取られます。因子が自動的に作成されているかを確認し、必要に応じて文字列形式に変換してください:
- 入力データの型を確認するには、
str(cats)を入力します。 - 出力で、コロンの後にある 3
文字のコードを確認します:
numとchrのみが表示される場合は、レッスンを続けることができます。このボックスはスキップしてください。fctが見つかった場合は、次の手順に進んでください。 - R
が因子データを自動的に作成しないようにするには、以下のコードを実行します:
options(stringsAsFactors = FALSE)。その後、catsテーブルを再読み込みして変更を反映させます。 - R を再起動するたびに、このオプションを設定する必要があります。忘れないように、データを読み込む前にスクリプトの最初の行のいずれかに含めてください。
- R バージョン 4.0.0 以降では、テキストデータは因子に変換されなくなりました。問題を回避するためにこのバージョン以降をインストールすることを検討してください。研究所や会社のコンピュータを使用している場合は、管理者に依頼してください。
データセットをすぐに探索し始めることができます。たとえば、$
演算子を使用して列を指定します:
R
cats$weight
OUTPUT
[1] 2.1 5.0 3.2R
cats$coat
OUTPUT
[1] "calico" "black" "tabby" 列に対して操作を実行することもできます:
R
## たとえば、スケールが 2kg 軽いことが判明した場合:
cats$weight + 2
OUTPUT
[1] 4.1 7.0 5.2R
paste("My cat is", cats$coat)
OUTPUT
[1] "My cat is calico" "My cat is black" "My cat is tabby" しかし、次のコードではどうでしょうか?
R
cats$weight + cats$coat
ERROR
Error in cats$weight + cats$coat: non-numeric argument to binary operatorここで何が起こったのかを理解することが、R でデータを成功裏に分析する鍵です。
データ型
最後のコマンドがエラーを返す理由が 2.1 と
"black"
を加算するのは無意味だからだと推測したなら、あなたは正しいです!これはプログラミングにおける重要な概念である
データ型
に関する直感をすでに持っているということです。データの型を調べるには、次のように入力します:
R
typeof(cats$weight)
OUTPUT
[1] "double"主なデータ型は次の 5
種類です:double、integer、complex、logical、character。
歴史的な理由で、double は numeric
とも呼ばれます。
R
typeof(3.14)
OUTPUT
[1] "double"R
typeof(1L) # L サフィックスを付けると数値を整数に強制します(R はデフォルトで浮動小数点数を使用)
OUTPUT
[1] "integer"R
typeof(1+1i)
OUTPUT
[1] "complex"R
typeof(TRUE)
OUTPUT
[1] "logical"R
typeof('banana')
OUTPUT
[1] "character"分析がどれだけ複雑であっても、R ではすべてのデータがこれらの基本的なデータ型のいずれかとして解釈されます。この厳格さには非常に重要な意味があります。
別の猫の詳細を追加した情報が、ファイル
data/feline-data_v2.csv に保存されています。
R
file.show("data/feline-data_v2.csv")
この新しい猫データを以前と同じ方法で読み込み、weight
列にどのようなデータ型が含まれているか確認します:
R
cats <- read.csv(file="data/feline-data_v2.csv")
typeof(cats
$weight)
OUTPUT
[1] "character"あらら、weight 列の型が double
ではなくなっています!以前と同じ計算を試みると、問題が発生します:
R
cats$weight + 2
ERROR
Error in cats$weight + 2: non-numeric argument to binary operator何が起こったのでしょうか? 私たちが扱っている cats
データは データフレーム と呼ばれるものです。データフレームは、R
で最も一般的で多用途な データ構造 の 1 つです。
データフレームの特定の列には異なるデータ型を混在させることはできません。
この場合、R はデータフレーム列 weight のすべてを
double
として読み取らなかったため、列全体のデータ型がその列内のすべてに適した型に変わります。
R が CSV ファイルを読み取ると、それは データフレーム
として読み込まれます。そのため、cats CSV
ファイルを読み込むと、データフレームとして保存されます。データフレームは
str() 関数によって表示される最初の行で認識できます:
R
str(cats)
OUTPUT
'data.frame': 4 obs. of 3 variables:
$ coat : chr "calico" "black" "tabby" "tabby"
$ weight : chr "2.1" "5" "3.2" "2.3 or 2.4"
$ likes_string: int 1 0 1 1データフレーム は行と列で構成され、各列は同じ数の行を持ちます。データフレームの異なる列は異なるデータ型で構成できます(これがデータフレームを非常に柔軟にする理由です)が、特定の列内ではすべてが同じ型である必要があります(例:ベクトル、因子、リストなど)。
この振る舞いをさらに調査する間、猫のデータから余分な行を削除し、それを再読み込みしましょう:
feline-data.csv:
coat,weight,likes_catnip
calico,2.1,1
black,5.0,0
tabby,3.2,1そして RStudio 内で:
R
cats <- read.csv(file="data/feline-data.csv")
ベクトルと型の強制変換
この挙動をよりよく理解するために、別のデータ構造である ベクトル を紹介します。
R
my_vector <- vector(length = 3)
my_vector
OUTPUT
[1] FALSE FALSE FALSER
におけるベクトルは、基本的に順序付けられた要素のリストです。ただし、特別な条件として、ベクトル内のすべての要素は同じ基本データ型である必要があります。データ型を指定しない場合、デフォルトで
logical
型になります。また、任意の型の空のベクトルを宣言することも可能です。
R
another_vector <- vector(mode='character', length=3)
another_vector
OUTPUT
[1] "" "" ""あるオブジェクトがベクトルかどうかを確認することもできます:
R
str(another_vector)
OUTPUT
chr [1:3] "" "" ""このコマンドのやや難解な出力は、このベクトルに含まれる基本データ型(この場合は
chr、文字型)を示し、ベクトル内の要素数(この場合は
[1:3])、および実際に含まれる要素(この場合は空の文字列)を示します。同様に次のコマンドを実行すると、
R
str(cats$weight)
OUTPUT
num [1:3] 2.1 5 3.2cats$weight もベクトルであることがわかります。R
のデータフレームに読み込まれる列はすべてベクトルです。これが、R
が列内のすべての要素を同じ基本データ型に強制する理由の根本です。
討論 1
なぜ R は列に含まれるデータに対してこれほど厳格なのでしょうか? この厳格さは私たちにどのように役立つのでしょうか?
列内のすべてのデータが同じであることで、データに対して単純な仮定を行うことができます。たとえば、列の 1 つの要素を数値として解釈できるなら、列内のすべての要素を数値として解釈できます。そのため、毎回確認する必要がなくなります。この一貫性こそが、人々が「クリーンデータ」と呼ぶものです。長い目で見ると、この厳格な一貫性は R におけるデータ操作を非常に簡単にしてくれます。
ベクトルを結合する際の型の強制変換
明示的な内容を持つベクトルを c()
関数で作成できます:
R
combine_vector <- c(2,6,3)
combine_vector
OUTPUT
[1] 2 6 3これまで学んだ内容を考えると、次のコードは何を生成すると思いますか?
R
quiz_vector <- c(2,6,'3')
これは 型の強制変換
と呼ばれるもので、予想外の結果をもたらすことがあり、基本データ型と R
がそれをどのように解釈するかを理解する必要があります。R
は、異なる型(ここでは double と
character)が単一のベクトルに結合される場合、それらをすべて同じ型に強制します。例を見てみましょう:
R
coercion_vector <- c('a', TRUE)
coercion_vector
OUTPUT
[1] "a" "TRUE"R
another_coercion_vector <- c(0, TRUE)
another_coercion_vector
OUTPUT
[1] 0 1型の階層
型の強制変換ルールは次の通りです:logical -> integer ->
double (“numeric”) -> complex
-> character
この矢印は「変換される」と読めます。たとえば、logical
と character を結合すると、結果は character
に変換されます:
R
c('a', TRUE)
OUTPUT
[1] "a" "TRUE"character
ベクトルは、印刷時にクォートで囲まれていることで簡単に認識できます。
逆方向の強制変換を試みる場合は、as.
関数を使用します:
R
character_vector_example <- c('0','2','4')
character_vector_example
OUTPUT
[1] "0" "2" "4"R
character_coerced_to_double <- as.double(character_vector_example)
character_coerced_to_double
OUTPUT
[1] 0 2 4R
double_coerced_to_logical <- as.logical(character_coerced_to_double)
double_coerced_to_logical
OUTPUT
[1] FALSE TRUE TRUER が基本データ型を他の型に強制する際に驚くべきことが起こる場合があります!型の強制変換の細かい点はさておき、重要なのは:データが予想していた形式と異なる場合、それは型の強制変換が原因である可能性が高いです。ベクトルやデータフレームの列内のすべてのデータが同じ型であることを確認してください。さもなければ、予想外の問題が発生する可能性があります!
しかし、強制変換は非常に便利な場合もあります!たとえば、cats
データの likes_catnip 列は数値型ですが、実際には 1 と 0
がそれぞれ TRUE と FALSE
を表しています。このデータには logical
型を使用すべきです。この型は TRUE または FALSE
の 2 状態を持ち、データの意味に完全に一致します。この列を
logical に「強制変換」するには、as.logical
関数を使用します:
R
cats$likes_catnip
OUTPUT
[1] 1 0 1R
cats$likes_catnip <- as.logical(cats$likes_catnip)
cats$likes_catnip
OUTPUT
[1] TRUE FALSE TRUEチャレンジ 1
データ分析の重要な部分は、入力データのクリーンアップです。入力データがすべて同じ形式(例:数値)であることを知っていると、分析がはるかに簡単になります!型の強制変換に関する章で扱った猫のデータセットをクリーンアップしましょう。
コードテンプレートをコピー
RStudio で新しいスクリプトを作成し、以下のコードをコピー&ペーストしてください。その後、以下のタスクを参考にギャップ(______)を埋めてください。
# データを読み込み
cats <- read.csv("data/feline-data_v2.csv")
# 1. データを表示
_____
# 2. 表の概要をデータ型と共に表示
_____(cats)
# 3. "weight" 列の現在のデータ型 __________。
# 正しいデータ型は: ____________。
# 4. 4 番目の "weight" データポイントを指定された 2 つの値の平均に修正
cats$weight[4] <- 2.35
# 効果を確認するためにデータを再表示
cats
# 5. "weight" を正しいデータ型に変換
cats$weight <- ______________(cats$weight)
# 自分でテストするために平均を計算
mean(cats$weight)
# 正しい平均値(NA ではない)が表示されたら、演習は完了です!2. データ型の概要を表示する
データ型はデータ自体と同じくらい重要です。以前見た関数を使用して、cats
テーブルのすべての列のデータ型を表示します。
3. 必要
なデータ型はどれですか?
表示されるデータ型は、このデータ(猫の体重)には適していません。必要なデータ型はどれですか?
- なぜ
read.csv()関数は正しいデータ型を選ばなかったのでしょうか? - コメントのギャップに猫の体重に適したデータ型を埋めてください!
型の階層 のセクションに戻り、利用可能なデータ型を確認してください。
4. 問題のある値を修正する
問題のある 4 行目に新しい体重値を割り当てるコードが提供されています。実行する前に考えてみてください。この例のように数値を割り当てた後のデータ型はどうなりますか? 実行後にデータ型を確認して、自分の予測が正しいか確認してください。
5. 列 “weight” を正しいデータ型に変換する
猫の体重は数値です。しかし、列にはまだ適切なデータ型が設定されていません。この列を浮動小数点数に強制変換してください。
データ型を変換する関数は as.
で始まります。このスクリプトの上部で関数を確認するか、RStudio
のオートコンプリート機能を使用してください。 “as.”
と入力し、TAB キーを押します。
チャレンジ 1.5 の解答
歴史的な理由で、2 つの同義の関数があります:
R
cats$weight <- as.double(cats$weight)
cats$weight <- as.numeric(cats$weight)
基本的なベクトル関数
c()
関数を使用すると、既存のベクトルに新しい要素を追加することができます:
R
ab_vector <- c('a', 'b')
ab_vector
OUTPUT
[1] "a" "b"R
combine_example <- c(ab_vector, 'SWC')
combine_example
OUTPUT
[1] "a" "b" "SWC"また、数列を生成することも可能です:
R
mySeries <- 1:10
mySeries
OUTPUT
[1] 1 2 3 4 5 6 7 8 9 10R
seq(10)
OUTPUT
[1] 1 2 3 4 5 6 7 8 9 10R
seq(1, 10, by=0.1)
OUTPUT
[1] 1.0 1.1 1.2 1.3 1.4 1.5 1.6 1.7 1.8 1.9 2.0 2.1 2.2 2.3 2.4
[16] 2.5 2.6 2.7 2.8 2.9 3.0 3.1 3.2 3.3 3.4 3.5 3.6 3.7 3.8 3.9
[31] 4.0 4.1 4.2 4.3 4.4 4.5 4.6 4.7 4.8 4.9 5.0 5.1 5.2 5.3 5.4
[46] 5.5 5.6 5.7 5.8 5.9 6.0 6.1 6.2 6.3 6.4 6.5 6.6 6.7 6.8 6.9
[61] 7.0 7.1 7.2 7.3 7.4 7.5 7.6 7.7 7.8 7.9 8.0 8.1 8.2 8.3 8.4
[76] 8.5 8.6 8.7 8.8 8.9 9.0 9.1 9.2 9.3 9.4 9.5 9.6 9.7 9.8 9.9
[91] 10.0ベクトルについていくつかの質問をすることもできます:
R
sequence_example <- 20:25
head(sequence_example, n=2)
OUTPUT
[1] 20 21R
tail(sequence_example, n=4)
OUTPUT
[1] 22 23 24 25R
length(sequence_example)
OUTPUT
[1] 6R
typeof(sequence_example)
OUTPUT
[1] "integer"ベクトルの特定の要素を取得するには、角括弧記法を使用します:
R
first_element <- sequence_example[1]
first_element
OUTPUT
[1] 20特定の要素を変更するには、角括弧を矢印の右側に使用します:
R
sequence_example[1] <- 30
sequence_example
OUTPUT
[1] 30 21 22 23 24 25チャレンジ 2
1 から 26 までの数を含むベクトルを作成します。その後、このベクトルを 2 倍にします。
R
x <- 1:26
x <- x * 2
リスト
次に紹介するデータ構造は list
です。リストは他のデータ型よりもシンプルで、何でも入れることができるのが特徴です。ベクトルでは要素の基本データ型を統一する必要がありましたが、リストは異なるデータ型を持つことができます:
R
list_example <- list(1, "a", TRUE, 1+4i)
list_example
OUTPUT
[[1]]
[1] 1
[[2]]
[1] "a"
[[3]]
[1] TRUE
[[4]]
[1] 1+4istr()
を使用してオブジェクトの構造を表示すると、すべての要素のデータ型を確認できます:
R
str(list_example)
OUTPUT
List of 4
$ : num 1
$ : chr "a"
$ : logi TRUE
$ : cplx 1+4iリストの用途は何でしょうか?例えば、異なるデータ型を持つ関連データを整理できます。これは、Excel のスプレッドシートのように複数の表をまとめるのと似ています。他にも多くの用途があります。
次の章で、驚くかもしれない別の例を紹介します。
リストの特定の要素を取得するには 二重角括弧 を使用します:
R
list_example[[2]]
OUTPUT
[1] "a"リストの要素には 名前 を付けることもできます。名前を値の前に等号で指定します:
R
another_list <- list(title = "Numbers", numbers = 1:10, data = TRUE )
another_list
OUTPUT
$title
[1] "Numbers"
$numbers
[1] 1 2 3 4 5 6 7 8 9 10
$data
[1] TRUEこれにより 名前付きリスト が生成されます。これで新しいアクセス方法が追加されます!
R
another_list$title
OUTPUT
[1] "Numbers"名前
名前を使用すると、要素に意味を持たせることができます。これにより、データだけでなく説明情報も持つことができます。これはオブジェクトに貼り付けられるラベルのような メタデータ です。R ではこれは 属性 と呼ばれます。属性により、オブジェクトをさらに操作することが可能になります。ここでは、定義された名前で要素にアクセスすることができます。
名前を使用してベクトルやリストにアクセスする
名前付きリストの生成方法はすでに学びました。名前付きベクトルを生成する方法も非常に似ています。以前このような関数を見たことがあるはずです:
R
pizza_price <- c( pizzasubito = 5.64, pizzafresh = 6.60, callapizza = 4.50 )
しかし、要素の取得方法はリストとは異なります:
R
pizza_price["pizzasubito"]
OUTPUT
pizzasubito
5.64 リストのアプローチは機能しません:
R
pizza_price$pizzafresh
ERROR
Error in pizza_price$pizzafresh: $ operator is invalid for atomic vectorsこのエラーメッセージを覚えておくと役立ちます。同じようなエラーに遭遇することが多いですが、これはリストと勘違いしてベクトルの要素にアクセスしようとした場合に発生します。
名前の取得と変更
名前だけに興味がある場合は、names()
関数を使用します:
R
names(pizza_price)
OUTPUT
[1] "pizzasubito" "pizzafresh" "callapizza" ベクトルの要素にアクセスしたり変更したりする方法を学びました。同じことが名前についても可能です:
R
names(pizza_price)[3]
OUTPUT
[1] "callapizza"R
names(pizza_price)[3] <- "call-a-pizza"
pizza_price
OUTPUT
pizzasubito pizzafresh call-a-pizza
5.64 6.60 4.50 チャレンジ 3
-
pizza_priceの名前のデータ型は何ですか?str()またはtypeof()関数を使用して調べてください。
オブジェクトの名前を取得するには、その名前を names(...)
で囲みます。同様に、名前のデータ型を取得するには、全体をさらに
typeof(...) で囲みます:
typeof(names(pizza))または、コードをわかりやすくするために新しい変数を使用します:
n <- names(pizza)
typeof(n)チャレンジ 4
既存のベクトルやリストの一部の名前を変更する代わりに、オブジェクトのすべての名前を設定することも可能です。次のコード形式を使用します(すべての大文字部分を置き換えてください):
names( OBJECT ) <- CHARACTER_VECTORアルファベットの各文字に番号を割り当てるベクトルを作成しましょう!
- 1 から 26 の数列を持つ
letter_noというベクトルを作成します。 - R には
LETTERSという組み込みオブジェクトがあります。これは A から Z までの 26 文字を含むベクトルです。この 26 文字をletter_noの名前として設定します。 -
letter_no["B"]を呼び出して、値が 2 であることを確認してください!
letter_no <- 1:26 # or seq(1,26)
names(letter_no) <- LETTERS
letter_no["B"]データフレーム
このレッスンの冒頭でデータフレームについて簡単に触れましたが、それはデータの表形式を表しています。例として示した猫のデータフレームについては詳細に掘り下げていませんでした:
R
cats
OUTPUT
coat weight likes_catnip
1 calico 2.1 TRUE
2 black 5.0 FALSE
3 tabby 3.2 TRUEここで少し驚くべきことに気づくかもしれません。次のコマンドを実行してみましょう:
R
typeof(cats)
OUTPUT
[1] "list"データフレームが「内部的にはリストのように見える」ことがわかります。以前、リストについて次のように説明しました:
リストは異なる型のデータを整理するためのもの
データフレームの列は、それぞれが異なる型のベクトルであり、同じ表に属することで整理されています。
データフレームは実際にはベクトルのリストです。データフレームが特別なのは、すべてのベクトルが同じ長さでなければならない点です。
この「特別さ」はどのようにオブジェクトに組み込まれているのでしょうか?R がそれを単なるリストではなく、表として扱うのはなぜでしょう?
R
class(cats)
OUTPUT
[1] "data.frame"クラス は名前と同様に、オブジェクトに付加される属性です。この属性は、そのオブジェクトが人間にとって何を意味するのかを示します。
ここで疑問に思うかもしれません:なぜオブジェクトの型を判断するための関数がもう一つ必要なのでしょうか?すでに
typeof() がありますよね?typeof()
はオブジェクトがコンピュータ内でどのように構築されているかを教えてくれます。一方、class()
はオブジェクトの人間にとっての意味を示します。したがって、typeof()
の出力は R で固定されています(主に 5
種類のデータ型)が、class() の出力は R
パッケージによって多様で拡張可能です。
cats
の例では、整数型、倍精度数値型、論理型の変数が含まれています。すでに見たように、データフレームの各列はベクトルです:
R
cats$coat
OUTPUT
[1] "calico" "black" "tabby" R
cats[,1]
OUTPUT
[1] "calico" "black" "tabby" R
typeof(cats[,1])
OUTPUT
[1] "character"R
str(cats[,1])
OUTPUT
chr [1:3] "calico" "black" "tabby"一方、各行は異なる変数の観測値であり、それ自体がデータフレームであり、異なる型の要素で構成されることができます:
R
cats[1,]
OUTPUT
coat weight likes_catnip
1 calico 2.1 TRUER
typeof(cats[1,])
OUTPUT
[1] "list"R
str(cats[1,])
OUTPUT
'data.frame': 1 obs. of 3 variables:
$ coat : chr "calico"
$ weight : num 2.1
$ likes_catnip: logi TRUEチャレンジ 5
データフレームから変数、観測値、要素を取得する方法はいくつかあります:
cats[1]cats[[1]]cats$coatcats["coat"]cats[1, 1]cats[, 1]cats[1, ]
これらの例を試して、それぞれが何を返すのかを説明してください。
ヒント: 返されるものを調べるには、typeof()
関数を使用してください。
R
cats[1]
OUTPUT
coat
1 calico
2 black
3 tabbyデータフレームはベクトルのリストと考えられます。単一ブラケット
[1]
はリストの最初のスライスを別のリストとして返します。この場合、それはデータフレームの最初の列です。
R
cats[[1]]
OUTPUT
[1] "calico" "black" "tabby" 二重ブラケット [[1]]
はリスト項目の内容を返します。この場合、最初の列の内容である
character 型のベクトルです。
R
cats$coat
OUTPUT
[1] "calico" "black" "tabby" $
を使用して名前で項目にアクセスします。coat
はデータフレームの最初の列であり、character
型のベクトルです。
R
cats["coat"]
OUTPUT
coat
1 calico
2 black
3 tabby単一ブラケット ["coat"]
を使用し、インデックス番号の代わりに列名を指定します。例 1
と同様に、返されるオブジェクトは list です。
R
cats[1, 1]
OUTPUT
[1] "calico"単一ブラケットを使用し、行と列の座標を指定します。この場合、1 行目 1 列目の値が返されます。オブジェクトは character 型のベクトルです。
R
cats[, 1]
OUTPUT
[1] "calico" "black" "tabby" 前の例と同様に単一ブラケットを使用し、行と列の座標を指定しますが、行座標が指定されていません。この場合、R は欠損値をその列のすべての要素として解釈し、ベクトル として返します。
R
cats[1, ]
OUTPUT
coat weight likes_catnip
1 calico 2.1 TRUE再び単一ブラケットを使用し、行と列の座標を指定しますが、今回は列座標が指定されていません。返される値は 1 行目のすべての値を含む list です。
ヒント: データフレーム列の名前変更
データフレームには列名があり、names()
関数でアクセスできます:
R
names(cats)
OUTPUT
[1] "coat" "weight" "likes_catnip"cats の 2
番目の列の名前を変更したい場合は、names(cats) の 2
番目の要素に新しい名前を割り当てます:
R
names(cats)[2] <- "weight_kg"
cats
OUTPUT
coat weight_kg likes_catnip
1 calico 2.1 TRUE
2 black 5.0 FALSE
3 tabby 3.2 TRUE行列(Matrix)
最後に紹介するのは行列です。ゼロで満たされた行列を宣言してみましょう:
R
matrix_example <- matrix(0, ncol=6, nrow=3)
matrix_example
OUTPUT
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0 0 0 0 0 0
[2,] 0 0 0 0 0 0
[3,] 0 0 0 0 0 0行列を特別なものにしているのは dim() 属性です:
R
dim(matrix_example)
OUTPUT
[1] 3 6他のデータ構造と同様に、行列について質問することも可能です:
R
typeof(matrix_example)
OUTPUT
[1] "double"R
class(matrix_example)
OUTPUT
[1] "matrix" "array" R
str(matrix_example)
OUTPUT
num [1:3, 1:6] 0 0 0 0 0 0 0 0 0 0 ...R
nrow(matrix_example)
OUTPUT
[1] 3R
ncol(matrix_example)
OUTPUT
[1] 6チャレンジ 6
次のコードの結果はどうなるでしょうか?
R
length(matrix_example)
OUTPUT
[1] 18実行して確認してください。予想は当たりましたか?なぜそのような結果になるのでしょうか?
行列は次元属性を持つベクトルであるため、length
は行列内の要素の総数を返します:
R
matrix_example <- matrix(0, ncol=6, nrow=3)
length(matrix_example)
OUTPUT
[1] 18チャレンジ 7
1 から 50 の数値を含む、列数 5、行数 10 の行列を作成します。
デフォルトの動作として、この行列は列ごとに値が埋められますか、それとも行ごとですか?
その動作を変更する方法を調べてください。(ヒント:matrix
のドキュメントを参照)
R
x <- matrix(1:50, ncol=5, nrow=10)
x <- matrix(1:50, ncol=5, nrow=10, byrow = TRUE) # 行ごとに埋める
チャレンジ 8
このワークショップの次のセクションに対応する 2 つの要素を持つリストを作成します:
- データ型
- データ構造
各データ型およびデータ構造の名前を文字型ベクトルに格納してください。
R
dataTypes <- c('double', 'complex', 'integer', 'character', 'logical')
dataStructures <- c('data.frame', 'vector', 'list', 'matrix')
answer <- list(dataTypes, dataStructures)
チャレンジ 9
以下の行列の R 出力を考えてみてください:
OUTPUT
[,1] [,2]
[1,] 4 1
[2,] 9 5
[3,] 10 7この行列を作成するために使用された正しいコマンドはどれでしょうか?各コマンドを確認し、入力する前に正しいものを考えてください。
他のコマンドでどのような行列が作成されるかを考えてみてください。
matrix(c(4, 1, 9, 5, 10, 7), nrow = 3)matrix(c(4, 9, 10, 1, 5, 7), ncol = 2, byrow = TRUE)matrix(c(4, 9, 10, 1, 5, 7), nrow = 2)matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)
R
matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)
-
read.csvを使用して R で表形式データを読み取ります。 - R の基本データ型は、double、integer、complex、logical、character です。
- データフレームや行列のようなデータ構造は、リストやベクトルを基にし、いくつかの属性が追加されています。
Content from データフレームの操作
Last updated on 2025-07-01 | Edit this page
Estimated time: 30 minutes
Overview
Questions
- データフレームをどのように操作できますか?
Objectives
- 行や列を追加または削除する。
- 2 つのデータフレームを結合する。
- データフレームのサイズ、列のクラス、名前、最初の数行などの基本的なプロパティを表示する。
これまでに、R の基本的なデータ型とデータ構造について学びました。以降の作業は、それらのツールを操作することに集約されます。最も頻繁に登場するのは、CSV ファイルから情報を読み込んで作成するデータフレームです。このレッスンでは、データフレームの操作についてさらに学びます。
データフレームに列や行を追加する
データフレームの列はベクトルであるため、列全体でデータ型が一貫しています。そのため、新しい列を追加したい場合は、まず新しいベクトルを作成します:
R
age <- c(2, 3, 5)
cats
OUTPUT
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1これを列として追加するには、次のようにします:
R
cbind(cats, age)
OUTPUT
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5ただし、データフレームの行数と異なる要素数を持つベクトルを追加しようとすると失敗します:
R
age <- c(2, 3, 5, 12)
cbind(cats, age)
ERROR
Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 4R
age <- c(2, 3)
cbind(cats, age)
ERROR
Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 2なぜ失敗するのでしょうか?R は、新しい列の各行に 1 つの要素が必要だと考えています:
R
nrow(cats)
OUTPUT
[1] 3R
length(age)
OUTPUT
[1] 2したがって、nrow(cats) と length(age)
が等しい必要があります。新しいデータフレームを作成して、cats
に上書きしてみましょう。
R
age <- c(2, 3, 5)
cats <- cbind(cats, age)
次に、行を追加してみましょう。データフレームの行はリストであることを既に学びました:
R
newRow <- list("tortoiseshell", 3.3, TRUE, 9)
cats <- rbind(cats, newRow)
新しい行が正しく追加されたことを確認します。
R
cats
OUTPUT
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5
4 tortoiseshell 3.3 1 9行を削除する
データフレームに行や列を追加する方法を学びました。次に、行を削除する方法を見てみましょう。
R
cats
OUTPUT
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5
4 tortoiseshell 3.3 1 9最後の行を削除したデータフレームを取得するには:
R
cats[-4, ]
OUTPUT
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5コンマの後に何も指定しないことで、4 行目全体を削除することを示します。
複数の行を削除することもできます。たとえば、次のようにベクトル内に行番号を指定します:cats[c(-3,-4), ]
列を削除する
データフレームの列を削除することもできます。「age」列を削除する場合、変数番号またはインデックスを使用する方法があります。
R
cats[,-4]
OUTPUT
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1
4 tortoiseshell 3.3 1コンマの前に何も指定しないことで、すべての行を保持することを示します。
または、インデックス名と %in%
演算子を使用して列を削除することもできます。%in%
演算子は、左側の引数(ここでは cats
の名前)の各要素について「この要素は右側の引数に含まれますか?」と尋ねます。
R
drop <- names(cats) %in% c("age")
cats[,!drop]
OUTPUT
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1
4 tortoiseshell 3.3 1論理演算子(%in%
など)による部分集合化については、次のエピソードで詳しく説明します。詳細は
論理演算を使用した部分集合化
を参照してください。
データフレームの結合
データフレームにデータを追加する際に覚えておくべき重要な点は、列はベクトル、行はリストであることです。2
つのデータフレームを rbind
を使用して結合することもできます:
R
cats <- rbind(cats, cats)
cats
OUTPUT
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5
4 tortoiseshell 3.3 1 9
5 calico 2.1 1 2
6 black 5.0 0 3
7 tabby 3.2 1 5
8 tortoiseshell 3.3 1 9チャレンジ 1
次の構文を使用して、新しいデータフレームを R 内で作成できます:
R
df <- data.frame(id = c("a", "b", "c"),
x = 1:3,
y = c(TRUE, TRUE, FALSE))
以下の情報を持つデータフレームを作成してください:
- 名
- 姓
- ラッキーナンバー
次に、rbind
を使用して隣の人のエントリを追加します。最後に、cbind
を使用して「コーヒーブレイクの時間ですか?」という質問への各人の回答を含む列を追加してください。
R
df <- data.frame(first = c("Grace"),
last = c("Hopper"),
lucky_number = c(0))
df <- rbind(df, list("Marie", "Curie", 238) )
df <- cbind(df, coffeetime = c(TRUE, TRUE))
実用的な例
これまで、猫データを使ってデータフレーム操作の基本を学びました。次に、これらのスキルを使用して、より現実的なデータセットを扱います。以前ダウンロードした
gapminder データセットを読み込んでみましょう:
R
gapminder <- read.csv("data/gapminder_data.csv")
その他のヒント
タブ区切り値ファイル(.tsv)を扱う場合は、区切り文字として
"\\t"を指定するか、read.delim()を使用します。ファイルをインターネットから直接ダウンロードしてコンピュータの指定したローカルフォルダに保存するには、
download.file関数を使用できます。保存されたファイルをread.csv関数で読み込む例:
R
download.file("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/main/episodes/data/gapminder_data.csv", destfile = "data/gapminder_data.csv")
gapminder <- read.csv("data/gapminder_data.csv")
- また、ファイルパスの代わりに Web アドレスを
read.csvに指定して、ファイルを直接 R に読み込むこともできます。この場合、ローカルにファイルを保存する必要はありません。例:
R
gapminder <- read.csv("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/main/episodes/data/gapminder_data.csv")
readxl パッケージ を使用すると、Excel スプレッドシートをプレーンテキストに変換せずに直接読み込むことができます。
"stringsAsFactors"引数を使用すると、文字列を因子として読み込むか文字列として読み込むかを指定できます。R バージョン 4.0 以降では、デフォルトで文字列は文字型として読み込まれますが、古いバージョンでは因子として読み込まれるのがデフォルトでした。詳細は前のエピソードのコールアウトを参照してください。
gapminder
データセットを調べてみましょう。最初に行うべきことは、str
を使用してデータの構造を確認することです:
R
str(gapminder)
OUTPUT
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...gapminder
の構造を調べる別の方法として、summary
関数を使用します。この関数は R
のさまざまなオブジェクトで使用できます。データフレームの場合、summary
は各列の数値的、表形式、または記述的な概要を提供します。数値または整数型の列は記述統計(四分位数や平均値)で、文字列型の列はその長さ、クラス、モードで説明されます。
R
summary(gapminder)
OUTPUT
country year pop continent
Length:1704 Min. :1952 Min. :6.001e+04 Length:1704
Class :character 1st Qu.:1966 1st Qu.:2.794e+06 Class :character
Mode :character Median :1980 Median :7.024e+06 Mode :character
Mean :1980 Mean :2.960e+07
3rd Qu.:1993 3rd Qu.:1.959e+07
Max. :2007 Max. :1.319e+09
lifeExp gdpPercap
Min. :23.60 Min. : 241.2
1st Qu.:48.20 1st Qu.: 1202.1
Median :60.71 Median : 3531.8
Mean :59.47 Mean : 7215.3
3rd Qu.:70.85 3rd Qu.: 9325.5
Max. :82.60 Max. :113523.1 str や summary
関数と合わせて、typeof
関数を使ってデータフレームの個々の列を調べることもできます:
R
typeof(gapminder$year)
OUTPUT
[1] "integer"R
typeof(gapminder$country)
OUTPUT
[1] "character"R
str(gapminder$country)
OUTPUT
chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...データフレームの次元に関する情報も調べることができます。str(gapminder) の出力によると、gapminder には
6 つの変数の 1704
個の観測値があります。このことを覚えた上で、次のコードが何を返すか考えてみてください:
R
length(gapminder)
OUTPUT
[1] 6データフレームの長さが行数(1704)であると考えるのが妥当ですが、実際にはそうではありません。データフレームはベクトルや因子のリストで構成されていることを思い出してください:
R
typeof(gapminder)
OUTPUT
[1] "list"length が 6 を返した理由は、gapminder が 6
列のリストで構築されているためです。データセットの行数と列数を取得するには次のようにします:
R
nrow(gapminder)
OUTPUT
[1] 1704R
ncol(gapminder)
OUTPUT
[1] 6あるいは、両方を一度に取得するには:
R
dim(gapminder)
OUTPUT
[1] 1704 6すべての列のタイトルを調べることもできます。後でアクセスする際に便利です:
R
colnames(gapminder)
OUTPUT
[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"ここで、R が報告する構造が自分の直感や予想と一致しているかどうかを確認することが重要です。各列のデータ型が妥当かどうかを確認してください。そうでない場合は、これまで学んだ R のデータ解釈の仕組みや、一貫性の重要性に基づいて問題を解決する必要があります。
データ型や構造が合理的であることを確認したら、データの探索を開始しましょう。最初の数行を確認します:
R
head(gapminder)
OUTPUT
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134チャレンジ 2
データの最後の数行や中間のいくつかの行も確認するのが良い習慣です。これをどのように行いますか?
特に中間の行を探すのは難しくありませんが、ランダムな行をいくつか取得することもできます。これをどのようにコード化しますか?
最後の数行を確認するには、R に既にある関数を使用すれば簡単です:
R
tail(gapminder)
tail(gapminder, n = 15)
では、途中の任意の行を確認するにはどうすればよいでしょうか?
再現性のある分析を確保するために、コードをスクリプトファイルに保存し、後で再利用できるようにしましょう。
チャレンジ 3
File -> New File -> R Script に移動し、gapminder
データセットを読み込むための R スクリプトを作成します。このスクリプトを
scripts/
ディレクトリに保存し、バージョン管理に追加してください。
その後、source
関数を使用してスクリプトを実行します。ファイルパスを引数として指定するか、RStudio
の「Source」ボタンを押します。
source
関数はスクリプト内で別のスクリプトを使用するために使用できます。同じ種類のファイルを何度も読み込む必要がある場合、一度スクリプトとして保存すれば、以降はそれを繰り返し利用できます。
R
download.file("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/main/episodes/data/gapminder_data.csv", destfile = "data/gapminder_data.csv")
gapminder <- read.csv(file = "data/gapminder_data.csv")
データを gapminder
変数に読み込むには次のようにします:
R
source(file = "scripts/load-gapminder.R")
チャレンジ 4
str(gapminder)
の出力をもう一度読み、リストやベクトルについて学んだこと、および
colnames や dim
の出力を活用して、str
が表示する内容を説明してください。理解できない部分があれば、隣の人と相談してみてください。
オブジェクト gapminder
はデータフレームで、列は次のようになっています:
-
countryとcontinentは文字列(character)。 -
yearは整数型のベクトル。 -
pop、lifeExp、gdpPercapは数値型のベクトル。
- 新しい列をデータフレームに追加するには
cbind()を使用します。 - 新しい行をデータフレームに追加するには
rbind()を使用します。 - データフレームから行を削除します。
- データフレームの構造を理解するために、
str()、summary()、nrow()、ncol()、dim()、colnames()、head()、typeof()を使用します。 -
read.csv()を使用して CSV ファイルを読み込みます。 - データフレームの
length()が何を表しているのか理解します。
Content from データの部分集合化
Last updated on 2025-07-01 | Edit this page
Estimated time: 50 minutes
Overview
Questions
- R でデータの部分集合をどのように扱うことができますか?
Objectives
- ベクトル、因子、行列、リスト、データフレームの部分集合化を学ぶ
- インデックス、名前、比較演算を使って個々の要素や複数の要素を抽出する方法を理解する
- さまざまなデータ構造から要素をスキップまたは削除する方法を習得する
R には強力な部分集合化のための演算子が数多く用意されています。それらを習得することで、あらゆる種類のデータセットで複雑な操作を簡単に行えるようになります。
あらゆる種類のオブジェクトを部分集合化するために 6 つの異なる方法があり、データ構造ごとに 3 種類の部分集合化演算子があります。
では、R の基本となる数値ベクトルを使って始めましょう。
R
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
x
OUTPUT
a b c d e
5.4 6.2 7.1 4.8 7.5 アトミックベクトル
R では、文字列、数値、または論理値を含む単純なベクトルを アトミックベクトル と呼びます。これは、さらに単純化できないためです。
このようにダミーベクトルを作成しました。この中身をどのように取得するのでしょうか?
インデックスを使用した要素のアクセス
ベクトルの要素を抽出するには、対応するインデックスを指定します(1 から始まります):
R
x[1]
OUTPUT
a
5.4 R
x[4]
OUTPUT
d
4.8 角括弧 [] 演算子は関数であり、ベクトルや行列に対して「n
番目の要素を取得する」という意味を持ちます。
複数の要素を一度に取得することもできます:
R
x[c(1, 3)]
OUTPUT
a c
5.4 7.1 またはベクトルの一部(スライス)を取得することも可能です:
R
x[1:4]
OUTPUT
a b c d
5.4 6.2 7.1 4.8 :
演算子は左の値から右の値までの数値のシーケンスを生成します。
R
1:4
OUTPUT
[1] 1 2 3 4R
c(1, 2, 3, 4)
OUTPUT
[1] 1 2 3 4同じ要素を複数回取得することもできます:
R
x[c(1,1,3)]
OUTPUT
a a c
5.4 5.4 7.1 ベクトルの長さを超えるインデックスを指定すると、R は欠損値を返します:
R
x[6]
OUTPUT
<NA>
NA これは NA を含む長さ 1 のベクトルであり、名前も
NA です。
0 番目の要素を要求すると、空のベクトルが返されます:
R
x[0]
OUTPUT
named numeric(0)R のベクトルの番号付けは 1 から始まる
多くのプログラミング言語(C や Python など)では、ベクトルの最初の要素のインデックスは 0 です。一方、R では最初の要素は 1 です。
要素のスキップと削除
ベクトルのインデックスに負の数を使用すると、指定した要素を除くすべての要素が返されます:
R
x[-2]
OUTPUT
a c d e
5.4 7.1 4.8 7.5 複数の要素をスキップすることも可能です:
R
x[c(-1, -5)] # または x[-c(1,5)]
OUTPUT
b c d
6.2 7.1 4.8 ヒント: 演算の順序
ベクトルの一部をスキップしようとすると、新しい人はよく間違えます。例えば:
R
x[-1:3]
このコードは次のようなエラーを返します:
ERROR
Error in x[-1:3]: only 0's may be mixed with negative subscriptsこれは演算の順序が関係しています。:
は実際には関数であり、最初の引数を -1、2 番目の引数を
3
としてシーケンスを生成します:c(-1, 0, 1, 2, 3)。
正しい解決策は、この関数呼び出しを括弧で囲み、-
演算子を結果に適用することです:
R
x[-(1:3)]
OUTPUT
d e
4.8 7.5 ベクトルから要素を削除するには、結果を変数に再割り当てする必要があります:
R
x <- x[-4]
x
OUTPUT
a b c e
5.4 6.2 7.1 7.5 チャレンジ 1
次のコードが与えられた場合:
R
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
print(x)
OUTPUT
a b c d e
5.4 6.2 7.1 4.8 7.5 次の出力を生成する少なくとも 2 つの異なるコマンドを考え出してください:
OUTPUT
b c d
6.2 7.1 4.8 2 つの異なるコマンドを見つけたら、隣の人と比較してみてください。異なる戦略を持っていましたか?
R
x[2:4]
OUTPUT
b c d
6.2 7.1 4.8 R
x[-c(1,5)]
OUTPUT
b c d
6.2 7.1 4.8 R
x[c(2,3,4)]
OUTPUT
b c d
6.2 7.1 4.8 名前によるサブセット
インデックスではなく名前を使って要素を抽出することができます:
R
x <- c(a=5.4, b=6.2, c=7.1, d=4.8, e=7.5) # ベクトルにその場で名前を付ける
x[c("a", "c")]
OUTPUT
a c
5.4 7.1 これは、オブジェクトをサブセットする際に、通常より信頼性の高い方法です。
サブセット操作を連続して行う場合、要素の位置が変わることがありますが、名前は常に変わりません!
他の論理演算を用いたサブセット
論理ベクトルを使ってサブセットを取ることもできます:
R
x[c(FALSE, FALSE, TRUE, FALSE, TRUE)]
OUTPUT
c e
7.1 7.5 比較演算子(例:>、<、==)は論理ベクトルを生成するため、それを使ってベクトルを簡潔にサブセットできます。以下のステートメントは、前の例と同じ結果を返します。
R
x[x > 7]
OUTPUT
c e
7.1 7.5 このステートメントを分解すると、まず x>7
が評価されて論理ベクトル c(FALSE, FALSE, TRUE, FALSE, TRUE)
が生成され、それに基づいて x の要素が選択されます。
名前によるインデックス操作を模倣するには、==
を使うことができます(比較には = ではなく ==
を使用する必要がある点に注意):
R
x[names(x) == "a"]
OUTPUT
a
5.4 ヒント: 複数の論理条件の結合
複数の論理条件を結合したい場合があります。たとえば、特定の範囲内の寿命を持つアジアまたはヨーロッパに位置するすべての国を見つけたいとします。このような場合、R では論理ベクトルを結合するための以下の演算子を使用できます:
-
&(論理AND): 左右がともにTRUEの場合にTRUEを返します。 -
|(論理OR): 左右のいずれか、または両方がTRUEの場合にTRUEを返します。
& や | の代わりに
&& や ||
が使われることもありますが、これらはベクトルの最初の要素だけを見て残りを無視します。データ解析では、通常1文字の演算子(&
や
|)を使用し、2文字の演算子はプログラミング(ステートメントの実行を決定する際など)で使用してください。
-
!(論理NOT):TRUEをFALSEに、FALSEをTRUEに変換します。単一の条件(例:!TRUEはFALSEになる)や、ベクトル全体(例:!c(TRUE, FALSE)はc(FALSE, TRUE)になる)を否定できます。
また、単一のベクトル内の要素を比較するために、all(すべての要素が
TRUE の場合に TRUE を返す)や
any(1つ以上の要素が TRUE の場合に
TRUE を返す)関数を使用できます。
チャレンジ 2
以下のコードを用いて:
R
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
print(x)
OUTPUT
a b c d e
5.4 6.2 7.1 4.8 7.5 x から 4 より大きく 7
未満の値を返すサブセットコマンドを書いてください。
R
x_subset <- x[x<7 & x>4]
print(x_subset)
OUTPUT
a b d
5.4 6.2 4.8 ヒント: 重複する名前
1つのベクトル内で複数の要素が同じ名前を持つことも可能です(データフレームでは、列名が重複することはありますが、行名は一意である必要があります)。以下の例を考えてみてください:
R
x <- 1:3
x
OUTPUT
[1] 1 2 3R
names(x) <- c('a', 'a', 'a')
x
OUTPUT
a a a
1 2 3 R
x['a'] # 最初の値のみを返す
OUTPUT
a
1 R
x[names(x) == 'a'] # すべての値を返す
OUTPUT
a a a
1 2 3 ヒント: 演算子に関するヘルプを得る方法
演算子を引用符で囲むことで、ヘルプを検索できます:help("%in%") または ?"%in%"。
名前付き要素をスキップする
名前付き要素をスキップまたは削除するのは少し難しいです。文字列を否定してスキップしようとすると、R は「文字列を否定する方法が分からない」というやや分かりにくいエラーを出します:
R
x <- c(a=5.4, b=6.2, c=7.1, d=4.8, e=7.5) # もう一度その場でベクトルに名前を付ける
x[-"a"]
ERROR
Error in -"a": invalid argument to unary operatorしかし、!=(等しくない)演算子を使用して論理ベクトルを構築すれば、望む動作を実現できます:
R
x[names(x) != "a"]
OUTPUT
b c d e
6.2 7.1 4.8 7.5 複数の名前付きインデックスをスキップするのはさらに難しいです。例えば、"a"
と "c" を削除しようとして次のように試みます:
R
x[names(x)!=c("a","c")]
WARNING
Warning in names(x) != c("a", "c"): longer object length is not a multiple of
shorter object lengthOUTPUT
b c d e
6.2 7.1 4.8 7.5 R は 何か をしましたが、警告を出しており、それが示す通り
間違った結果 を返しました("c"
要素がまだベクトルに含まれています)。
!=
がこの場合に何を実際にしているのかは、非常に良い質問です。
リサイクル
このコードの比較部分を見てみましょう:
R
names(x) != c("a", "c")
WARNING
Warning in names(x) != c("a", "c"): longer object length is not a multiple of
shorter object lengthOUTPUT
[1] FALSE TRUE TRUE TRUE TRUEnames(x)[3] != "c" は明らかに偽なのに、なぜ R
はこのベクトルの3番目の要素に TRUE
を返すのでしょうか?!= を使用すると、R
は左辺の各要素を右辺の対応する要素と比較しようとします。左辺と右辺の長さが異なる場合はどうなりますか?
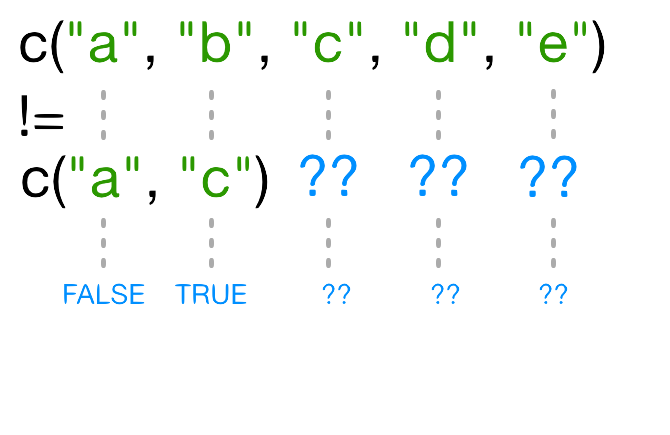
一方のベクトルがもう一方より短い場合、短いベクトルは リサイクル されます:
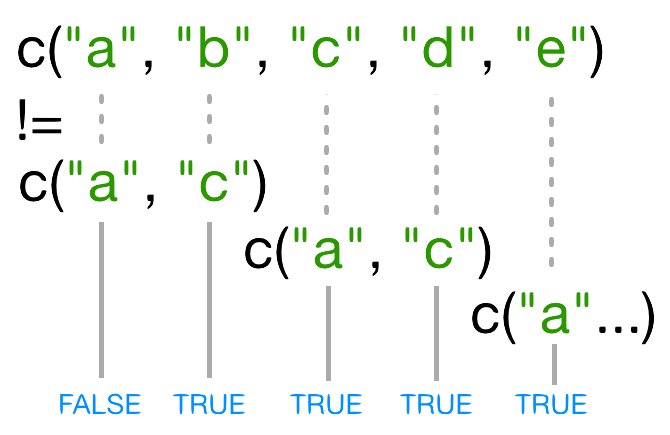
この場合、R は c("a", "c") を必要な回数だけ繰り返して
names(x)
と一致させます(例:c("a","c","a","c","a"))。
再利用された "a" が names(x)
の3番目の要素と一致しないため、!= の結果が
TRUE になります。
リサイクルによりこのような間違いが発生するのを防ぐには
%in%
演算子を使用します。この演算子は左辺の各要素について、右辺の中にその要素が存在するかどうかを確認します。今回は値を除外したいので、!
演算子も使用します:
R
x[! names(x) %in% c("a","c") ]
OUTPUT
b d e
6.2 4.8 7.5 チャレンジ 3
ベクトルの要素を、特定のリスト内のいずれかと一致させる操作は、データ解析で非常に一般的なタスクです。例えば、gapminder
データセットには country と continent
の変数がありますが、これらの間の情報は含まれていません。東南アジアの情報を抽出したいとします。このとき、どのようにして東南アジアのすべての国について
TRUE、それ以外を FALSE
とする論理ベクトルを作成しますか?
以下のデータを使用します:
R
seAsia <- c("Myanmar","Thailand","Cambodia","Vietnam","Laos")
## エピソード2でダウンロードした gapminder データを読み込む
gapminder <- read.csv("data/gapminder_data.csv", header=TRUE)
## データフレームから `country` 列を抽出(詳細は後述);
## factor を文字列に変換;
## 重複しない要素のみ取得
countries <- unique(as.character(gapminder$country))
以下の3つの方法を試し、それぞれがどのように(正しくない、または正しい方法で)動作するのか説明してください:
-
間違った方法(
==のみを使用)
-
不格好な方法(論理演算子
==と|を使用)
-
エレガントな方法(
%in%を使用)
間違った方法
countries==seAsia
この方法では、警告("In countries == seAsia : 長いオブジェクトの長さが短いオブジェクトの長さの倍数ではありません")が表示され、誤った結果(すべてFALSEのベクトル)が返されます。これは、seAsiaの再利用された値が正しい位置に一致しないためです。不格好な方法
以下のコードでは正しい値を得ることができますが、非常に冗長で扱いにくいです:
R
(countries=="Myanmar" | countries=="Thailand" |
countries=="Cambodia" | countries == "Vietnam" | countries=="Laos")
または、countries==seAsia[1] | countries==seAsia[2] | ...
のように記述します。
リストが長い場合、さらに複雑になります。
-
エレガントな方法
countries %in% seAsia
この方法は正確で、記述が簡単で可読性も高いです。
特殊値の扱い
R
では、欠損値、無限値、未定義値を処理できない関数に出会うことがあります。
そのようなデータをフィルタリングするために、以下の特殊な関数を使用できます:
-
is.na:ベクトル、行列、またはデータフレーム内のNA(またはNaN)を含む位置を返します。 - 同様に、
is.nanとis.infiniteは、それぞれNaNとInfに対応します。 -
is.finite:NA、NaN、Infを含まない位置を返します。 -
na.omit:ベクトルからすべての欠損値を除外します。
因子のサブセット
ベクトルのサブセット方法を学んだところで、他のデータ構造のサブセットについて考えてみましょう。
因子のサブセットは、ベクトルのサブセットと同じ方法で行えます。
R
f <- factor(c("a", "a", "b", "c", "c", "d"))
f[f == "a"]
OUTPUT
[1] a a
Levels: a b c dR
f[f %in% c("b", "c")]
OUTPUT
[1] b c c
Levels: a b c dR
f[1:3]
OUTPUT
[1] a a b
Levels: a b c d要素をスキップしても、そのカテゴリが因子レベルから削除されるわけではありません:
R
f[-3]
OUTPUT
[1] a a c c d
Levels: a b c d行列のサブセット
行列は [
関数を使用してサブセットします。この場合、2つの引数を取り、1つ目は行、2つ目は列に適用されます:
R
set.seed(1)
m <- matrix(rnorm(6*4), ncol=4, nrow=6)
m[3:4, c(3,1)]
OUTPUT
[,1] [,2]
[1,] 1.12493092 -0.8356286
[2,] -0.04493361 1.5952808行または列全体を取得する場合は、1つ目または2つ目の引数を空白のままにします:
R
m[, c(3,4)]
OUTPUT
[,1] [,2]
[1,] -0.62124058 0.82122120
[2,] -2.21469989 0.59390132
[3,] 1.12493092 0.91897737
[4,] -0.04493361 0.78213630
[5,] -0.01619026 0.07456498
[6,] 0.94383621 -1.989351701つの行または列のみを取得すると、結果が自動的にベクトルに変換されます:
R
m[3,]
OUTPUT
[1] -0.8356286 0.5757814 1.1249309 0.9189774結果を行列として保持するには、第3引数 を指定して
drop = FALSE を設定します:
R
m[3, , drop=FALSE]
OUTPUT
[,1] [,2] [,3] [,4]
[1,] -0.8356286 0.5757814 1.124931 0.9189774行または列の外側をアクセスしようとすると、R はエラーをスローします:
R
m[, c(3,6)]
ERROR
Error in m[, c(3, 6)]: subscript out of boundsヒント: 高次元配列
多次元配列の場合、[
の各引数が次元に対応します。例えば、3次元配列の場合、最初の3つの引数が行、列、および深さ次元に対応します。
行列はベクトルであるため、1つの引数だけを使用してサブセットを取ることもできます:
R
m[5]
OUTPUT
[1] 0.3295078これは通常あまり有用ではなく、読み取りづらい場合があります。ただし、行列がデフォルトで 列優先フォーマット に配置されていることを理解するのに役立ちます。つまり、ベクトルの要素は列ごとに配置されます:
R
matrix(1:6, nrow=2, ncol=3)
OUTPUT
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6行ごとに行列を埋めたい場合は、byrow=TRUE
を使用します:
R
matrix(1:6, nrow=2, ncol=3, byrow=TRUE)
OUTPUT
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6行列は、行および列のインデックスの代わりに、その行名および列名を使用してサブセットを取ることもできます。
チャレンジ 4
以下のコードを用いて:
R
m <- matrix(1:18, nrow=3, ncol=6)
print(m)
OUTPUT
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 1 4 7 10 13 16
[2,] 2 5 8 11 14 17
[3,] 3 6 9 12 15 18- 以下のコマンドのうち、11 と 14 を抽出するものはどれでしょうか?
A. m[2,4,2,5]
B. m[2:5]
C. m[4:5,2]
D. m[2,c(4,5)]
D
リストのサブセット
ここでは新しいサブセット演算子を紹介します。リストをサブセットするためには、3つの関数を使用します。これらは、原子ベクトルや行列を学ぶ際にも登場しました:[,
[[, $ です。
[
を使用すると、常にリストが返されます。リストから要素を「抽出」するのではなく「サブセット」したい場合に使用します。
R
xlist <- list(a = "Software Carpentry", b = 1:10, data = head(mtcars))
xlist[1]
OUTPUT
$a
[1] "Software Carpentry"このコードは、1つの要素を含むリスト を返します。
リストの要素は、原子ベクトルと同じ方法でサブセットできます。ただし、比較演算は再帰的ではなく、リスト内のデータ構造に基づいて条件が評価されるため、リスト内の個々の要素には適用されません。
R
xlist[1:2]
OUTPUT
$a
[1] "Software Carpentry"
$b
[1] 1 2 3 4 5 6 7 8 9 10リストの個々の要素を抽出するには、二重角括弧関数 [[
を使用する必要があります。
R
xlist[[1]]
OUTPUT
[1] "Software Carpentry"この結果はリストではなくベクトルであることに注意してください。
複数の要素を一度に抽出することはできません:
R
xlist[[1:2]]
ERROR
Error in xlist[[1:2]]: subscript out of boundsまた、要素をスキップすることもできません:
R
xlist[[-1]]
ERROR
Error in xlist[[-1]]: invalid negative subscript in get1index <real>ただし、名前を使用して要素をサブセットおよび抽出することは可能です:
R
xlist[["a"]]
OUTPUT
[1] "Software Carpentry"$
演算子は、名前で要素を抽出するための簡潔な記法を提供します:
R
xlist$data
OUTPUT
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1チャレンジ 5
以下のリストが与えられています:
R
xlist <- list(a = "Software Carpentry", b = 1:10, data = head(mtcars))
リストとベクトルのサブセット方法を用いて、xlist
から数字の 2 を抽出してください。
ヒント:数字の 2 はリスト内の "b" に含まれています。
R
xlist$b[2]
OUTPUT
[1] 2R
xlist[[2]][2]
OUTPUT
[1] 2R
xlist[["b"]][2]
OUTPUT
[1] 2チャレンジ 6
以下の線形モデルが与えられています:
R
mod <- aov(pop ~ lifeExp, data=gapminder)
残差の自由度を抽出してください(ヒント:attributes()
が役立ちます)。
R
attributes(mod) ## `df.residual` は `mod` の名前の1つです
R
mod$df.residual
データフレーム
データフレームは内部的にはリストであることを覚えておきましょう。そのため、同様のルールが適用されます。ただし、データフレームは2次元のオブジェクトでもあります:
[
に1つの引数を与える場合、リストと同様に動作し、それぞれのリスト要素が列に対応します。結果として得られるオブジェクトはデータフレームになります:
R
head(gapminder[3])
OUTPUT
pop
1 8425333
2 9240934
3 10267083
4 11537966
5 13079460
6 14880372同様に、[[ を使用すると、単一の列
を抽出します:
R
head(gapminder[["lifeExp"]])
OUTPUT
[1] 28.801 30.332 31.997 34.020 36.088 38.438$
演算子は、列を名前で抽出するための便利なショートカットを提供します:
R
head(gapminder$year)
OUTPUT
[1] 1952 1957 1962 1967 1972 19772つの引数を与えると、[
は行列と同じように動作します:
R
gapminder[1:3,]
OUTPUT
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007単一行をサブセットすると、結果はデータフレームになります(要素が混合型のためです):
R
gapminder[3,]
OUTPUT
country year pop continent lifeExp gdpPercap
3 Afghanistan 1962 10267083 Asia 31.997 853.1007ただし、単一列をサブセットすると結果はベクトルになります(第3引数
drop = FALSE を指定することで変更可能)。
チャレンジ 7
以下の一般的なデータフレームサブセットエラーを修正してください:
- 年 1957 の観測値を抽出する
- 1列目から4列目以外のすべての列を抽出する
R
gapminder[,-1:4]
- 寿命が80年以上の行を抽出する
R
gapminder[gapminder$lifeExp > 80]
- 1行目と4列目、5列目(
continentとlifeExp)を抽出する
R
gapminder[1, 4, 5]
- 応用:年 2002 年と 2007 年の情報を含む行を抽出する
R
gapminder[gapminder$year == 2002 | 2007,]
以下の一般的なデータフレームサブセットエラーを修正:
- 年 1957 の観測値を抽出する
R
# gapminder[gapminder$year = 1957,]
gapminder[gapminder$year == 1957,]
- 1列目から4列目以外のすべての列を抽出する
R
# gapminder[,-1:4]
gapminder[,-c(1:4)]
- 寿命が80年以上の行を抽出する
R
# gapminder[gapminder$lifeExp > 80]
gapminder[gapminder$lifeExp > 80,]
- 1行目と4列目、5列目(
continentとlifeExp)を抽出する
R
# gapminder[1, 4, 5]
gapminder[1, c(4, 5)]
- 応用:年 2002 年と 2007 年の情報を含む行を抽出する
R
# gapminder[gapminder$year == 2002 | 2007,]
gapminder[gapminder$year == 2002 | gapminder$year == 2007,]
gapminder[gapminder$year %in% c(2002, 2007),]
Challenge 8
Why does
gapminder[1:20]return an error? How does it differ fromgapminder[1:20, ]?Create a new
data.framecalledgapminder_smallthat only contains rows 1 through 9 and 19 through 23. You can do this in one or two steps.
gapminderis a data.frame so needs to be subsetted on two dimensions.gapminder[1:20, ]subsets the data to give the first 20 rows and all columns.
R
gapminder_small <- gapminder[c(1:9, 19:23),]
- Indexing in R starts at 1, not 0.
- Access individual values by location using
[]. - Access slices of data using
[low:high]. - Access arbitrary sets of data using
[c(...)]. - Use logical operations and logical vectors to access subsets of data.
Content from Control Flow
Last updated on 2025-07-01 | Edit this page
Estimated time: 65 minutes
Overview
Questions
- How can I make data-dependent choices in R?
- How can I repeat operations in R?
Objectives
- Write conditional statements with
if...elsestatements andifelse(). - Write and understand
for()loops.
Often when we’re coding we want to control the flow of our actions. This can be done by setting actions to occur only if a condition or a set of conditions are met. Alternatively, we can also set an action to occur a particular number of times.
There are several ways you can control flow in R. For conditional statements, the most commonly used approaches are the constructs:
R
# if
if (condition is true) {
perform action
}
# if ... else
if (condition is true) {
perform action
} else { # that is, if the condition is false,
perform alternative action
}Say, for example, that we want R to print a message if a variable
x has a particular value:
R
x <- 8
if (x >= 10) {
print("x is greater than or equal to 10")
}
x
OUTPUT
[1] 8The print statement does not appear in the console because x is not
greater than 10. To print a different message for numbers less than 10,
we can add an else statement.
R
x <- 8
if (x >= 10) {
print("x is greater than or equal to 10")
} else {
print("x is less than 10")
}
OUTPUT
[1] "x is less than 10"You can also test multiple conditions by using
else if.
R
x <- 8
if (x >= 10) {
print("x is greater than or equal to 10")
} else if (x > 5) {
print("x is greater than 5, but less than 10")
} else {
print("x is less than 5")
}
OUTPUT
[1] "x is greater than 5, but less than 10"Important: when R evaluates the condition inside
if() statements, it is looking for a logical element, i.e.,
TRUE or FALSE. This can cause some headaches
for beginners. For example:
R
x <- 4 == 3
if (x) {
"4 equals 3"
} else {
"4 does not equal 3"
}
OUTPUT
[1] "4 does not equal 3"As we can see, the not equal message was printed because the vector x
is FALSE
R
x <- 4 == 3
x
OUTPUT
[1] FALSEChallenge 1
Use an if() statement to print a suitable message
reporting whether there are any records from 2002 in the
gapminder dataset. Now do the same for 2012.
We will first see a solution to Challenge 1 which does not use the
any() function. We first obtain a logical vector describing
which element of gapminder$year is equal to
2002:
R
gapminder[(gapminder$year == 2002),]
Then, we count the number of rows of the data.frame
gapminder that correspond to the 2002:
R
rows2002_number <- nrow(gapminder[(gapminder$year == 2002),])
The presence of any record for the year 2002 is equivalent to the
request that rows2002_number is one or more:
R
rows2002_number >= 1
Putting all together, we obtain:
R
if(nrow(gapminder[(gapminder$year == 2002),]) >= 1){
print("Record(s) for the year 2002 found.")
}
All this can be done more quickly with any(). The
logical condition can be expressed as:
R
if(any(gapminder$year == 2002)){
print("Record(s) for the year 2002 found.")
}
Did anyone get a warning message like this?
ERROR
Error in if (gapminder$year == 2012) {: the condition has length > 1The if() function only accepts singular (of length 1)
inputs, and therefore returns an error when you use it with a vector.
The if() function will still run, but will only evaluate
the condition in the first element of the vector. Therefore, to use the
if() function, you need to make sure your input is singular
(of length 1).
Tip: Built in ifelse()
function
R accepts both if() and
else if() statements structured as outlined above, but also
statements using R’s built-in ifelse()
function. This function accepts both singular and vector inputs and is
structured as follows:
where the first argument is the condition or a set of conditions to
be met, the second argument is the statement that is evaluated when the
condition is TRUE, and the third statement is the statement
that is evaluated when the condition is FALSE.
R
y <- -3
ifelse(y < 0, "y is a negative number", "y is either positive or zero")
OUTPUT
[1] "y is a negative number"Tip: any() and
all()
The any() function will return TRUE if at
least one TRUE value is found within a vector, otherwise it
will return FALSE. This can be used in a similar way to the
%in% operator. The function all(), as the name
suggests, will only return TRUE if all values in the vector
are TRUE.
Repeating operations
If you want to iterate over a set of values, when the order of
iteration is important, and perform the same operation on each, a
for() loop will do the job. We saw for() loops
in the shell
lessons earlier. This is the most flexible of looping operations,
but therefore also the hardest to use correctly. In general, the advice
of many R users would be to learn about for()
loops, but to avoid using for() loops unless the order of
iteration is important: i.e. the calculation at each iteration depends
on the results of previous iterations. If the order of iteration is not
important, then you should learn about vectorized alternatives, such as
the purrr package, as they pay off in computational
efficiency.
The basic structure of a for() loop is:
For example:
R
for (i in 1:10) {
print(i)
}
OUTPUT
[1] 1
[1] 2
[1] 3
[1] 4
[1] 5
[1] 6
[1] 7
[1] 8
[1] 9
[1] 10The 1:10 bit creates a vector on the fly; you can
iterate over any other vector as well.
We can use a for() loop nested within another
for() loop to iterate over two things at once.
R
for (i in 1:5) {
for (j in c('a', 'b', 'c', 'd', 'e')) {
print(paste(i,j))
}
}
OUTPUT
[1] "1 a"
[1] "1 b"
[1] "1 c"
[1] "1 d"
[1] "1 e"
[1] "2 a"
[1] "2 b"
[1] "2 c"
[1] "2 d"
[1] "2 e"
[1] "3 a"
[1] "3 b"
[1] "3 c"
[1] "3 d"
[1] "3 e"
[1] "4 a"
[1] "4 b"
[1] "4 c"
[1] "4 d"
[1] "4 e"
[1] "5 a"
[1] "5 b"
[1] "5 c"
[1] "5 d"
[1] "5 e"We notice in the output that when the first index (i) is
set to 1, the second index (j) iterates through its full
set of indices. Once the indices of j have been iterated
through, then i is incremented. This process continues
until the last index has been used for each for() loop.
Rather than printing the results, we could write the loop output to a new object.
R
output_vector <- c()
for (i in 1:5) {
for (j in c('a', 'b', 'c', 'd', 'e')) {
temp_output <- paste(i, j)
output_vector <- c(output_vector, temp_output)
}
}
output_vector
OUTPUT
[1] "1 a" "1 b" "1 c" "1 d" "1 e" "2 a" "2 b" "2 c" "2 d" "2 e" "3 a" "3 b"
[13] "3 c" "3 d" "3 e" "4 a" "4 b" "4 c" "4 d" "4 e" "5 a" "5 b" "5 c" "5 d"
[25] "5 e"This approach can be useful, but ‘growing your results’ (building the result object incrementally) is computationally inefficient, so avoid it when you are iterating through a lot of values.
Tip: don’t grow your results
One of the biggest things that trips up novices and experienced R users alike, is building a results object (vector, list, matrix, data frame) as your for loop progresses. Computers are very bad at handling this, so your calculations can very quickly slow to a crawl. It’s much better to define an empty results object before hand of appropriate dimensions, rather than initializing an empty object without dimensions. So if you know the end result will be stored in a matrix like above, create an empty matrix with 5 row and 5 columns, then at each iteration store the results in the appropriate location.
A better way is to define your (empty) output object before filling in the values. For this example, it looks more involved, but is still more efficient.
R
output_matrix <- matrix(nrow = 5, ncol = 5)
j_vector <- c('a', 'b', 'c', 'd', 'e')
for (i in 1:5) {
for (j in 1:5) {
temp_j_value <- j_vector[j]
temp_output <- paste(i, temp_j_value)
output_matrix[i, j] <- temp_output
}
}
output_vector2 <- as.vector(output_matrix)
output_vector2
OUTPUT
[1] "1 a" "2 a" "3 a" "4 a" "5 a" "1 b" "2 b" "3 b" "4 b" "5 b" "1 c" "2 c"
[13] "3 c" "4 c" "5 c" "1 d" "2 d" "3 d" "4 d" "5 d" "1 e" "2 e" "3 e" "4 e"
[25] "5 e"Tip: While loops
Sometimes you will find yourself needing to repeat an operation as
long as a certain condition is met. You can do this with a
while() loop.
R will interpret a condition being met as “TRUE”.
As an example, here’s a while loop that generates random numbers from
a uniform distribution (the runif() function) between 0 and
1 until it gets one that’s less than 0.1.
R
z <- 1
while(z > 0.1){
z <- runif(1)
cat(z, "\n")
}
while() loops will not always be appropriate. You have
to be particularly careful that you don’t end up stuck in an infinite
loop because your condition is always met and hence the while statement
never terminates.
Challenge 2
Compare the objects output_vector and
output_vector2. Are they the same? If not, why not? How
would you change the last block of code to make
output_vector2 the same as output_vector?
We can check whether the two vectors are identical using the
all() function:
R
all(output_vector == output_vector2)
However, all the elements of output_vector can be found
in output_vector2:
R
all(output_vector %in% output_vector2)
and vice versa:
R
all(output_vector2 %in% output_vector)
therefore, the element in output_vector and
output_vector2 are just sorted in a different order. This
is because as.vector() outputs the elements of an input
matrix going over its column. Taking a look at
output_matrix, we can notice that we want its elements by
rows. The solution is to transpose the output_matrix. We
can do it either by calling the transpose function t() or
by inputting the elements in the right order. The first solution
requires to change the original
R
output_vector2 <- as.vector(output_matrix)
into
R
output_vector2 <- as.vector(t(output_matrix))
The second solution requires to change
R
output_matrix[i, j] <- temp_output
into
R
output_matrix[j, i] <- temp_output
Challenge 3
Write a script that loops through the gapminder data by
continent and prints out whether the mean life expectancy is smaller or
larger than 50 years.
Step 1: We want to make sure we can extract all the unique values of the continent vector
R
gapminder <- read.csv("data/gapminder_data.csv")
unique(gapminder$continent)
Step 2: We also need to loop over each of these
continents and calculate the average life expectancy for each
subset of data. We can do that as follows:
- Loop over each of the unique values of ‘continent’
- For each value of continent, create a temporary variable storing that subset
- Return the calculated life expectancy to the user by printing the output:
R
for (iContinent in unique(gapminder$continent)) {
tmp <- gapminder[gapminder$continent == iContinent, ]
cat(iContinent, mean(tmp$lifeExp, na.rm = TRUE), "\n")
rm(tmp)
}
Step 3: The exercise only wants the output printed
if the average life expectancy is less than 50 or greater than 50. So we
need to add an if() condition before printing, which
evaluates whether the calculated average life expectancy is above or
below a threshold, and prints an output conditional on the result. We
need to amend (3) from above:
3a. If the calculated life expectancy is less than some threshold (50 years), return the continent and a statement that life expectancy is less than threshold, otherwise return the continent and a statement that life expectancy is greater than threshold:
R
thresholdValue <- 50
for (iContinent in unique(gapminder$continent)) {
tmp <- mean(gapminder[gapminder$continent == iContinent, "lifeExp"])
if (tmp < thresholdValue){
cat("Average Life Expectancy in", iContinent, "is less than", thresholdValue, "\n")
} else {
cat("Average Life Expectancy in", iContinent, "is greater than", thresholdValue, "\n")
} # end if else condition
rm(tmp)
} # end for loop
Challenge 4
Modify the script from Challenge 3 to loop over each country. This time print out whether the life expectancy is smaller than 50, between 50 and 70, or greater than 70.
We modify our solution to Challenge 3 by now adding two thresholds,
lowerThreshold and upperThreshold and
extending our if-else statements:
R
lowerThreshold <- 50
upperThreshold <- 70
for (iCountry in unique(gapminder$country)) {
tmp <- mean(gapminder[gapminder$country == iCountry, "lifeExp"])
if(tmp < lowerThreshold) {
cat("Average Life Expectancy in", iCountry, "is less than", lowerThreshold, "\n")
} else if(tmp > lowerThreshold && tmp < upperThreshold) {
cat("Average Life Expectancy in", iCountry, "is between", lowerThreshold, "and", upperThreshold, "\n")
} else {
cat("Average Life Expectancy in", iCountry, "is greater than", upperThreshold, "\n")
}
rm(tmp)
}
Challenge 5 - Advanced
Write a script that loops over each country in the
gapminder dataset, tests whether the country starts with a
‘B’, and graphs life expectancy against time as a line graph if the mean
life expectancy is under 50 years.
We will use the grep() command that was introduced in
the Unix
Shell lesson to find countries that start with “B.” Lets understand
how to do this first. Following from the Unix shell section we may be
tempted to try the following
R
grep("^B", unique(gapminder$country))
But when we evaluate this command it returns the indices of the
factor variable country that start with “B.” To get the
values, we must add the value=TRUE option to the
grep() command:
R
grep("^B", unique(gapminder$country), value = TRUE)
We will now store these countries in a variable called
candidateCountries, and then loop over each entry in the variable.
Inside the loop, we evaluate the average life expectancy for each
country, and if the average life expectancy is less than 50 we use
base-plot to plot the evolution of average life expectancy using
with() and subset():
R
thresholdValue <- 50
candidateCountries <- grep("^B", unique(gapminder$country), value = TRUE)
for (iCountry in candidateCountries) {
tmp <- mean(gapminder[gapminder$country == iCountry, "lifeExp"])
if (tmp < thresholdValue) {
cat("Average Life Expectancy in", iCountry, "is less than", thresholdValue, "plotting life expectancy graph... \n")
with(subset(gapminder, country == iCountry),
plot(year, lifeExp,
type = "o",
main = paste("Life Expectancy in", iCountry, "over time"),
ylab = "Life Expectancy",
xlab = "Year"
) # end plot
) # end with
} # end if
rm(tmp)
} # end for loop
- Use
ifandelseto make choices. - Use
forto repeat operations.
Content from Creating Publication-Quality Graphics with ggplot2
Last updated on 2025-07-01 | Edit this page
Estimated time: 80 minutes
Overview
Questions
- How can I create publication-quality graphics in R?
Objectives
- To be able to use ggplot2 to generate publication-quality graphics.
- To apply geometry, aesthetic, and statistics layers to a ggplot plot.
- To manipulate the aesthetics of a plot using different colors, shapes, and lines.
- To improve data visualization through transforming scales and paneling by group.
- To save a plot created with ggplot to disk.
Plotting our data is one of the best ways to quickly explore it and the various relationships between variables.
There are three main plotting systems in R, the base plotting system, the lattice package, and the ggplot2 package.
Today we’ll be learning about the ggplot2 package, because it is the most effective for creating publication-quality graphics.
ggplot2 is built on the grammar of graphics, the idea that any plot can be built from the same set of components: a data set, mapping aesthetics, and graphical layers:
Data sets are the data that you, the user, provide.
Mapping aesthetics are what connect the data to the graphics. They tell ggplot2 how to use your data to affect how the graph looks, such as changing what is plotted on the X or Y axis, or the size or color of different data points.
Layers are the actual graphical output from ggplot2. Layers determine what kinds of plot are shown (scatterplot, histogram, etc.), the coordinate system used (rectangular, polar, others), and other important aspects of the plot. The idea of layers of graphics may be familiar to you if you have used image editing programs like Photoshop, Illustrator, or Inkscape.
Let’s start off building an example using the gapminder data from
earlier. The most basic function is ggplot, which lets R
know that we’re creating a new plot. Any of the arguments we give the
ggplot function are the global options for the
plot: they apply to all layers on the plot.
R
library("ggplot2")
ggplot(data = gapminder)

Here we called ggplot and told it what data we want to
show on our figure. This is not enough information for
ggplot to actually draw anything. It only creates a blank
slate for other elements to be added to.
Now we’re going to add in the mapping aesthetics
using the aes function. aes tells
ggplot how variables in the data map to
aesthetic properties of the figure, such as which columns of
the data should be used for the x and
y locations.
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp))
Here we told ggplot we want to plot the “gdpPercap”
column of the gapminder data frame on the x-axis, and the “lifeExp”
column on the y-axis. Notice that we didn’t need to explicitly pass
aes these columns
(e.g. x = gapminder[, "gdpPercap"]), this is because
ggplot is smart enough to know to look in the
data for that column!
The final part of making our plot is to tell ggplot how
we want to visually represent the data. We do this by adding a new
layer to the plot using one of the
geom functions.
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()
Here we used geom_point, which tells ggplot
we want to visually represent the relationship between
x and y as a scatterplot of
points.
Challenge 1
Modify the example so that the figure shows how life expectancy has changed over time:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) + geom_point()
Hint: the gapminder dataset has a column called “year”, which should appear on the x-axis.
Here is one possible solution:
R
ggplot(data = gapminder, mapping = aes(x = year, y = lifeExp)) + geom_point()
Challenge 2
In the previous examples and challenge we’ve used the
aes function to tell the scatterplot geom
about the x and y locations of each
point. Another aesthetic property we can modify is the point
color. Modify the code from the previous challenge to
color the points by the “continent” column. What trends
do you see in the data? Are they what you expected?
The solution presented below adds color=continent to the
call of the aes function. The general trend seems to
indicate an increased life expectancy over the years. On continents with
stronger economies we find a longer life expectancy.
R
ggplot(data = gapminder, mapping = aes(x = year, y = lifeExp, color=continent)) +
geom_point()
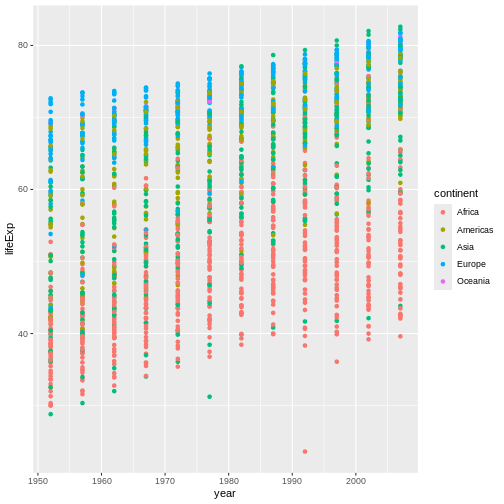
Layers
Using a scatterplot probably isn’t the best for visualizing change
over time. Instead, let’s tell ggplot to visualize the data
as a line plot:
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, color=continent)) +
geom_line()
Instead of adding a geom_point layer, we’ve added a
geom_line layer.
However, the result doesn’t look quite as we might have expected: it seems to be jumping around a lot in each continent. Let’s try to separate the data by country, plotting one line for each country:
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, group=country, color=continent)) +
geom_line()
We’ve added the group aesthetic, which
tells ggplot to draw a line for each country.
But what if we want to visualize both lines and points on the plot? We can add another layer to the plot:
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, group=country, color=continent)) +
geom_line() + geom_point()
It’s important to note that each layer is drawn on top of the previous layer. In this example, the points have been drawn on top of the lines. Here’s a demonstration:
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, group=country)) +
geom_line(mapping = aes(color=continent)) + geom_point()
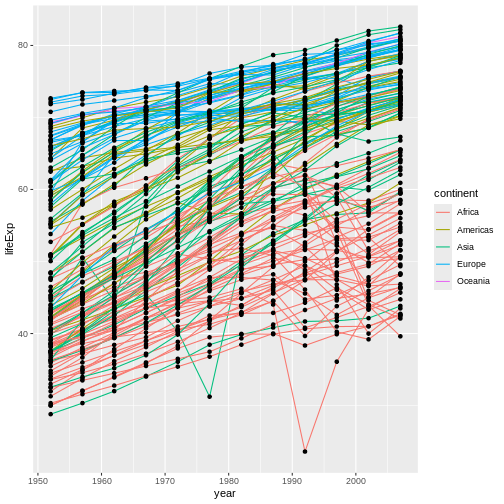
In this example, the aesthetic mapping of
color has been moved from the global plot options in
ggplot to the geom_line layer so it no longer
applies to the points. Now we can clearly see that the points are drawn
on top of the lines.
Tip: Setting an aesthetic to a value instead of a mapping
So far, we’ve seen how to use an aesthetic (such as
color) as a mapping to a variable in the data.
For example, when we use
geom_line(mapping = aes(color=continent)), ggplot will give
a different color to each continent. But what if we want to change the
color of all lines to blue? You may think that
geom_line(mapping = aes(color="blue")) should work, but it
doesn’t. Since we don’t want to create a mapping to a specific variable,
we can move the color specification outside of the aes()
function, like this: geom_line(color="blue").
Challenge 3
Switch the order of the point and line layers from the previous example. What happened?
The lines now get drawn over the points!
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, group=country)) +
geom_point() + geom_line(mapping = aes(color=continent))
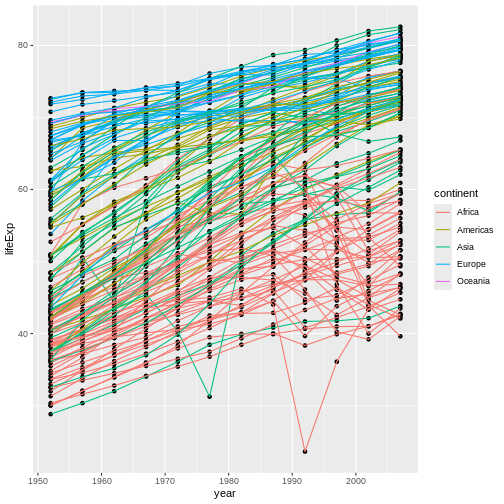
Transformations and statistics
ggplot2 also makes it easy to overlay statistical models over the data. To demonstrate we’ll go back to our first example:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()
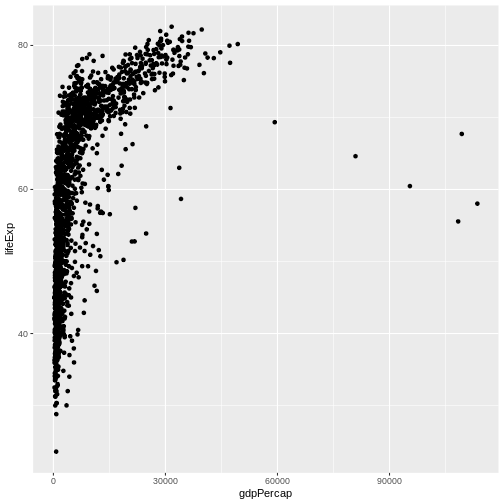
Currently it’s hard to see the relationship between the points due to some strong outliers in GDP per capita. We can change the scale of units on the x axis using the scale functions. These control the mapping between the data values and visual values of an aesthetic. We can also modify the transparency of the points, using the alpha function, which is especially helpful when you have a large amount of data which is very clustered.
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10()
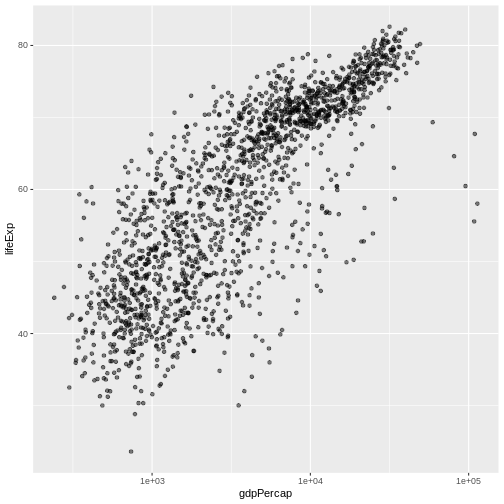
The scale_x_log10 function applied a transformation to
the coordinate system of the plot, so that each multiple of 10 is evenly
spaced from left to right. For example, a GDP per capita of 1,000 is the
same horizontal distance away from a value of 10,000 as the 10,000 value
is from 100,000. This helps to visualize the spread of the data along
the x-axis.
Tip Reminder: Setting an aesthetic to a value instead of a mapping
Notice that we used geom_point(alpha = 0.5). As the
previous tip mentioned, using a setting outside of the
aes() function will cause this value to be used for all
points, which is what we want in this case. But just like any other
aesthetic setting, alpha can also be mapped to a variable in
the data. For example, we can give a different transparency to each
continent with
geom_point(mapping = aes(alpha = continent)).
We can fit a simple relationship to the data by adding another layer,
geom_smooth:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10() + geom_smooth(method="lm")
OUTPUT
`geom_smooth()` using formula = 'y ~ x'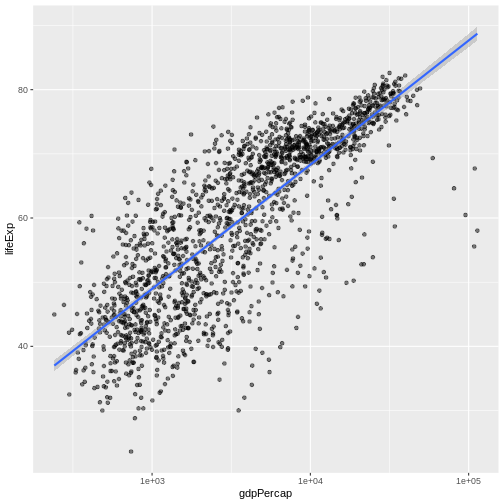
We can make the line thicker by setting the
linewidth aesthetic in the geom_smooth
layer:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10() + geom_smooth(method="lm", linewidth=1.5)
OUTPUT
`geom_smooth()` using formula = 'y ~ x'
There are two ways an aesthetic can be specified. Here we
set the linewidth aesthetic by passing it as
an argument to geom_smooth and it is applied the same to
the whole geom. Previously in the lesson we’ve used the
aes function to define a mapping between data
variables and their visual representation.
Challenge 4a
Modify the color and size of the points on the point layer in the previous example.
Hint: do not use the aes function.
Hint: the equivalent of linewidth for points is
size.
Here a possible solution: Notice that the color argument
is supplied outside of the aes() function. This means that
it applies to all data points on the graph and is not related to a
specific variable.
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(size=3, color="orange") + scale_x_log10() +
geom_smooth(method="lm", linewidth=1.5)
OUTPUT
`geom_smooth()` using formula = 'y ~ x'
Challenge 4b
Modify your solution to Challenge 4a so that the points are now a different shape and are colored by continent with new trendlines. Hint: The color argument can be used inside the aesthetic.
Here is a possible solution: Notice that supplying the
color argument inside the aes() functions
enables you to connect it to a certain variable. The shape
argument, as you can see, modifies all data points the same way (it is
outside the aes() call) while the color
argument which is placed inside the aes() call modifies a
point’s color based on its continent value.
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp, color = continent)) +
geom_point(size=3, shape=17) + scale_x_log10() +
geom_smooth(method="lm", linewidth=1.5)
OUTPUT
`geom_smooth()` using formula = 'y ~ x'
Multi-panel figures
Earlier we visualized the change in life expectancy over time across all countries in one plot. Alternatively, we can split this out over multiple panels by adding a layer of facet panels.
Tip
We start by making a subset of data including only countries located in the Americas. This includes 25 countries, which will begin to clutter the figure. Note that we apply a “theme” definition to rotate the x-axis labels to maintain readability. Nearly everything in ggplot2 is customizable.
R
americas <- gapminder[gapminder$continent == "Americas",]
ggplot(data = americas, mapping = aes(x = year, y = lifeExp)) +
geom_line() +
facet_wrap( ~ country) +
theme(axis.text.x = element_text(angle = 45))
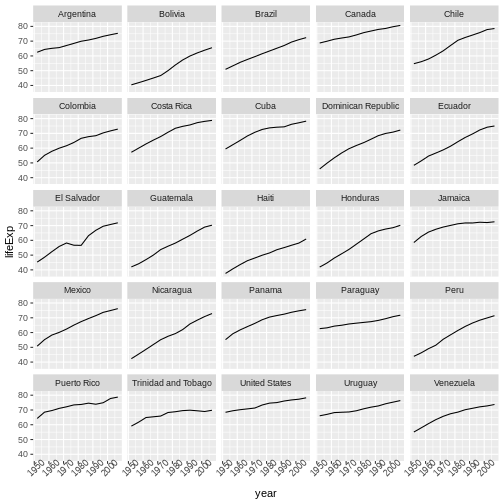
The facet_wrap layer took a “formula” as its argument,
denoted by the tilde (~). This tells R to draw a panel for each unique
value in the country column of the gapminder dataset.
Modifying text
To clean this figure up for a publication we need to change some of the text elements. The x-axis is too cluttered, and the y axis should read “Life expectancy”, rather than the column name in the data frame.
We can do this by adding a couple of different layers. The
theme layer controls the axis text, and overall text
size. Labels for the axes, plot title and any legend can be set using
the labs function. Legend titles are set using the same
names we used in the aes specification. Thus below the
color legend title is set using color = "Continent", while
the title of a fill legend would be set using
fill = "MyTitle".
R
ggplot(data = americas, mapping = aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country) +
labs(
x = "Year", # x axis title
y = "Life expectancy", # y axis title
title = "Figure 1", # main title of figure
color = "Continent" # title of legend
) +
theme(axis.text.x = element_text(angle = 90, hjust = 1))
Exporting the plot
The ggsave() function allows you to export a plot
created with ggplot. You can specify the dimension and resolution of
your plot by adjusting the appropriate arguments (width,
height and dpi) to create high quality
graphics for publication. In order to save the plot from above, we first
assign it to a variable lifeExp_plot, then tell
ggsave to save that plot in png format to a
directory called results. (Make sure you have a
results/ folder in your working directory.)
R
lifeExp_plot <- ggplot(data = americas, mapping = aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country) +
labs(
x = "Year", # x axis title
y = "Life expectancy", # y axis title
title = "Figure 1", # main title of figure
color = "Continent" # title of legend
) +
theme(axis.text.x = element_text(angle = 90, hjust = 1))
ggsave(filename = "results/lifeExp.png", plot = lifeExp_plot, width = 12, height = 10, dpi = 300, units = "cm")
There are two nice things about ggsave. First, it
defaults to the last plot, so if you omit the plot argument
it will automatically save the last plot you created with
ggplot. Secondly, it tries to determine the format you want
to save your plot in from the file extension you provide for the
filename (for example .png or .pdf). If you
need to, you can specify the format explicitly in the
device argument.
This is a taste of what you can do with ggplot2. RStudio provides a really useful cheat sheet of the different layers available, and more extensive documentation is available on the ggplot2 website. All RStudio cheat sheets are available from the RStudio website. Finally, if you have no idea how to change something, a quick Google search will usually send you to a relevant question and answer on Stack Overflow with reusable code to modify!
Challenge 5
Generate boxplots to compare life expectancy between the different continents during the available years.
Advanced:
- Rename y axis as Life Expectancy.
- Remove x axis labels.
Here a possible solution: xlab() and ylab()
set labels for the x and y axes, respectively The axis title, text and
ticks are attributes of the theme and must be modified within a
theme() call.
R
ggplot(data = gapminder, mapping = aes(x = continent, y = lifeExp, fill = continent)) +
geom_boxplot() + facet_wrap(~year) +
ylab("Life Expectancy") +
theme(axis.title.x=element_blank(),
axis.text.x = element_blank(),
axis.ticks.x = element_blank())
- Use
ggplot2to create plots. - Think about graphics in layers: aesthetics, geometry, statistics, scale transformation, and grouping.
Content from Vectorization
Last updated on 2025-07-01 | Edit this page
Estimated time: 25 minutes
Overview
Questions
- How can I operate on all the elements of a vector at once?
Objectives
- To understand vectorized operations in R.
Most of R’s functions are vectorized, meaning that the function will operate on all elements of a vector without needing to loop through and act on each element one at a time. This makes writing code more concise, easy to read, and less error prone.
R
x <- 1:4
x * 2
OUTPUT
[1] 2 4 6 8The multiplication happened to each element of the vector.
We can also add two vectors together:
R
y <- 6:9
x + y
OUTPUT
[1] 7 9 11 13Each element of x was added to its corresponding element
of y:
Here is how we would add two vectors together using a for loop:
R
output_vector <- c()
for (i in 1:4) {
output_vector[i] <- x[i] + y[i]
}
output_vector
OUTPUT
[1] 7 9 11 13Compare this to the output using vectorised operations.
R
sum_xy <- x + y
sum_xy
OUTPUT
[1] 7 9 11 13Challenge 1
Let’s try this on the pop column of the
gapminder dataset.
Make a new column in the gapminder data frame that
contains population in units of millions of people. Check the head or
tail of the data frame to make sure it worked.
Let’s try this on the pop column of the
gapminder dataset.
Make a new column in the gapminder data frame that
contains population in units of millions of people. Check the head or
tail of the data frame to make sure it worked.
R
gapminder$pop_millions <- gapminder$pop / 1e6
head(gapminder)
OUTPUT
country year pop continent lifeExp gdpPercap pop_millions
1 Afghanistan 1952 8425333 Asia 28.801 779.4453 8.425333
2 Afghanistan 1957 9240934 Asia 30.332 820.8530 9.240934
3 Afghanistan 1962 10267083 Asia 31.997 853.1007 10.267083
4 Afghanistan 1967 11537966 Asia 34.020 836.1971 11.537966
5 Afghanistan 1972 13079460 Asia 36.088 739.9811 13.079460
6 Afghanistan 1977 14880372 Asia 38.438 786.1134 14.880372Challenge 2
On a single graph, plot population, in millions, against year, for all countries. Do not worry about identifying which country is which.
Repeat the exercise, graphing only for China, India, and Indonesia. Again, do not worry about which is which.
Refresh your plotting skills by plotting population in millions against year.
R
ggplot(gapminder, aes(x = year, y = pop_millions)) +
geom_point()
R
countryset <- c("China","India","Indonesia")
ggplot(gapminder[gapminder$country %in% countryset,],
aes(x = year, y = pop_millions)) +
geom_point()
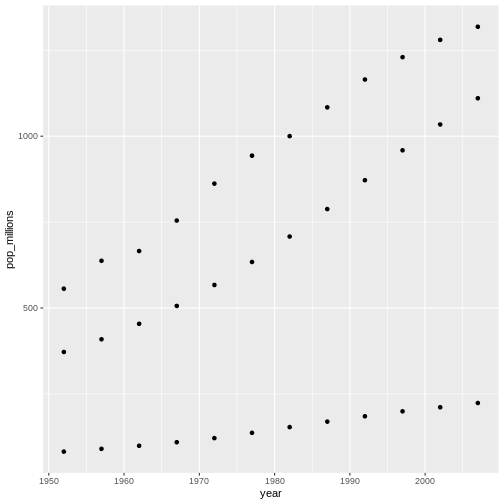
Comparison operators, logical operators, and many functions are also vectorized:
Comparison operators
R
x > 2
OUTPUT
[1] FALSE FALSE TRUE TRUELogical operators
R
a <- x > 3 # or, for clarity, a <- (x > 3)
a
OUTPUT
[1] FALSE FALSE FALSE TRUETip: some useful functions for logical vectors
any() will return TRUE if any
element of a vector is TRUE.all() will return TRUE if all
elements of a vector are TRUE.
Most functions also operate element-wise on vectors:
Functions
R
x <- 1:4
log(x)
OUTPUT
[1] 0.0000000 0.6931472 1.0986123 1.3862944Vectorized operations work element-wise on matrices:
R
m <- matrix(1:12, nrow=3, ncol=4)
m * -1
OUTPUT
[,1] [,2] [,3] [,4]
[1,] -1 -4 -7 -10
[2,] -2 -5 -8 -11
[3,] -3 -6 -9 -12Tip: element-wise vs. matrix multiplication
Very important: the operator * gives you element-wise
multiplication! To do matrix multiplication, we need to use the
%*% operator:
R
m %*% matrix(1, nrow=4, ncol=1)
OUTPUT
[,1]
[1,] 22
[2,] 26
[3,] 30R
matrix(1:4, nrow=1) %*% matrix(1:4, ncol=1)
OUTPUT
[,1]
[1,] 30For more on matrix algebra, see the Quick-R reference guide
Challenge 3
Given the following matrix:
R
m <- matrix(1:12, nrow=3, ncol=4)
m
OUTPUT
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12Write down what you think will happen when you run:
m ^ -1m * c(1, 0, -1)m > c(0, 20)m * c(1, 0, -1, 2)
Did you get the output you expected? If not, ask a helper!
Given the following matrix:
R
m <- matrix(1:12, nrow=3, ncol=4)
m
OUTPUT
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12Write down what you think will happen when you run:
m ^ -1
OUTPUT
[,1] [,2] [,3] [,4]
[1,] 1.0000000 0.2500000 0.1428571 0.10000000
[2,] 0.5000000 0.2000000 0.1250000 0.09090909
[3,] 0.3333333 0.1666667 0.1111111 0.08333333m * c(1, 0, -1)
OUTPUT
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 0 0 0 0
[3,] -3 -6 -9 -12m > c(0, 20)
OUTPUT
[,1] [,2] [,3] [,4]
[1,] TRUE FALSE TRUE FALSE
[2,] FALSE TRUE FALSE TRUE
[3,] TRUE FALSE TRUE FALSEChallenge 4
We’re interested in looking at the sum of the following sequence of fractions:
R
x = 1/(1^2) + 1/(2^2) + 1/(3^2) + ... + 1/(n^2)
This would be tedious to type out, and impossible for high values of n. Use vectorisation to compute x when n=100. What is the sum when n=10,000?
We’re interested in looking at the sum of the following sequence of fractions:
R
x = 1/(1^2) + 1/(2^2) + 1/(3^2) + ... + 1/(n^2)
This would be tedious to type out, and impossible for high values of n. Can you use vectorisation to compute x, when n=100? How about when n=10,000?
R
sum(1/(1:100)^2)
OUTPUT
[1] 1.634984R
sum(1/(1:1e04)^2)
OUTPUT
[1] 1.644834R
n <- 10000
sum(1/(1:n)^2)
OUTPUT
[1] 1.644834We can also obtain the same results using a function:
R
inverse_sum_of_squares <- function(n) {
sum(1/(1:n)^2)
}
inverse_sum_of_squares(100)
OUTPUT
[1] 1.634984R
inverse_sum_of_squares(10000)
OUTPUT
[1] 1.644834R
n <- 10000
inverse_sum_of_squares(n)
OUTPUT
[1] 1.644834Tip: Operations on vectors of unequal length
Operations can also be performed on vectors of unequal length, through a process known as recycling. This process automatically repeats the smaller vector until it matches the length of the larger vector. R will provide a warning if the larger vector is not a multiple of the smaller vector.
R
x <- c(1, 2, 3)
y <- c(1, 2, 3, 4, 5, 6, 7)
x + y
WARNING
Warning in x + y: longer object length is not a multiple of shorter object
lengthOUTPUT
[1] 2 4 6 5 7 9 8Vector x was recycled to match the length of vector
y
- Use vectorized operations instead of loops.
Content from Functions Explained
Last updated on 2025-07-01 | Edit this page
Estimated time: 60 minutes
Overview
Questions
- How can I write a new function in R?
Objectives
- Define a function that takes arguments.
- Return a value from a function.
- Check argument conditions with
stopifnot()in functions. - Test a function.
- Set default values for function arguments.
- Explain why we should divide programs into small, single-purpose functions.
If we only had one data set to analyze, it would probably be faster to load the file into a spreadsheet and use that to plot simple statistics. However, the gapminder data is updated periodically, and we may want to pull in that new information later and re-run our analysis again. We may also obtain similar data from a different source in the future.
In this lesson, we’ll learn how to write a function so that we can repeat several operations with a single command.
What is a function?
Functions gather a sequence of operations into a whole, preserving it for ongoing use. Functions provide:
- a name we can remember and invoke it by
- relief from the need to remember the individual operations
- a defined set of inputs and expected outputs
- rich connections to the larger programming environment
As the basic building block of most programming languages, user-defined functions constitute “programming” as much as any single abstraction can. If you have written a function, you are a computer programmer.
Defining a function
Let’s open a new R script file in the functions/
directory and call it functions-lesson.R.
The general structure of a function is:
R
my_function <- function(parameters) {
# perform action
# return value
}
Let’s define a function fahr_to_kelvin() that converts
temperatures from Fahrenheit to Kelvin:
R
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
We define fahr_to_kelvin() by assigning it to the output
of function. The list of argument names are contained
within parentheses. Next, the body of
the function–the statements that are executed when it runs–is contained
within curly braces ({}). The statements in the body are
indented by two spaces. This makes the code easier to read but does not
affect how the code operates.
It is useful to think of creating functions like writing a cookbook. First you define the “ingredients” that your function needs. In this case, we only need one ingredient to use our function: “temp”. After we list our ingredients, we then say what we will do with them, in this case, we are taking our ingredient and applying a set of mathematical operators to it.
When we call the function, the values we pass to it as arguments are assigned to those variables so that we can use them inside the function. Inside the function, we use a return statement to send a result back to whoever asked for it.
Tip
One feature unique to R is that the return statement is not required. R automatically returns whichever variable is on the last line of the body of the function. But for clarity, we will explicitly define the return statement.
Let’s try running our function. Calling our own function is no different from calling any other function:
R
# freezing point of water
fahr_to_kelvin(32)
OUTPUT
[1] 273.15R
# boiling point of water
fahr_to_kelvin(212)
OUTPUT
[1] 373.15Challenge 1
Write a function called kelvin_to_celsius() that takes a
temperature in Kelvin and returns that temperature in Celsius.
Hint: To convert from Kelvin to Celsius you subtract 273.15
Write a function called kelvin_to_celsius that takes a
temperature in Kelvin and returns that temperature in Celsius
R
kelvin_to_celsius <- function(temp) {
celsius <- temp - 273.15
return(celsius)
}
Combining functions
The real power of functions comes from mixing, matching and combining them into ever-larger chunks to get the effect we want.
Let’s define two functions that will convert temperature from Fahrenheit to Kelvin, and Kelvin to Celsius:
R
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
kelvin_to_celsius <- function(temp) {
celsius <- temp - 273.15
return(celsius)
}
Challenge 2
Define the function to convert directly from Fahrenheit to Celsius, by reusing the two functions above (or using your own functions if you prefer).
Define the function to convert directly from Fahrenheit to Celsius, by reusing these two functions above
R
fahr_to_celsius <- function(temp) {
temp_k <- fahr_to_kelvin(temp)
result <- kelvin_to_celsius(temp_k)
return(result)
}
Interlude: Defensive Programming
Now that we’ve begun to appreciate how writing functions provides an
efficient way to make R code re-usable and modular, we should note that
it is important to ensure that functions only work in their intended
use-cases. Checking function parameters is related to the concept of
defensive programming. Defensive programming encourages us to
frequently check conditions and throw an error if something is wrong.
These checks are referred to as assertion statements because we want to
assert some condition is TRUE before proceeding. They make
it easier to debug because they give us a better idea of where the
errors originate.
Checking conditions with stopifnot()
Let’s start by re-examining fahr_to_kelvin(), our
function for converting temperatures from Fahrenheit to Kelvin. It was
defined like so:
R
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
For this function to work as intended, the argument temp
must be a numeric value; otherwise, the mathematical
procedure for converting between the two temperature scales will not
work. To create an error, we can use the function stop().
For example, since the argument temp must be a
numeric vector, we could check for this condition with an
if statement and throw an error if the condition was
violated. We could augment our function above like so:
R
fahr_to_kelvin <- function(temp) {
if (!is.numeric(temp)) {
stop("temp must be a numeric vector.")
}
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
If we had multiple conditions or arguments to check, it would take
many lines of code to check all of them. Luckily R provides the
convenience function stopifnot(). We can list as many
requirements that should evaluate to TRUE;
stopifnot() throws an error if it finds one that is
FALSE. Listing these conditions also serves a secondary
purpose as extra documentation for the function.
Let’s try out defensive programming with stopifnot() by
adding assertions to check the input to our function
fahr_to_kelvin().
We want to assert the following: temp is a numeric
vector. We may do that like so:
R
fahr_to_kelvin <- function(temp) {
stopifnot(is.numeric(temp))
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
It still works when given proper input.
R
# freezing point of water
fahr_to_kelvin(temp = 32)
OUTPUT
[1] 273.15But fails instantly if given improper input.
R
# Metric is a factor instead of numeric
fahr_to_kelvin(temp = as.factor(32))
ERROR
Error in fahr_to_kelvin(temp = as.factor(32)): is.numeric(temp) is not TRUEChallenge 3
Use defensive programming to ensure that our
fahr_to_celsius() function throws an error immediately if
the argument temp is specified inappropriately.
Extend our previous definition of the function by adding in an
explicit call to stopifnot(). Since
fahr_to_celsius() is a composition of two other functions,
checking inside here makes adding checks to the two component functions
redundant.
R
fahr_to_celsius <- function(temp) {
stopifnot(is.numeric(temp))
temp_k <- fahr_to_kelvin(temp)
result <- kelvin_to_celsius(temp_k)
return(result)
}
More on combining functions
Now, we’re going to define a function that calculates the Gross Domestic Product of a nation from the data available in our dataset:
R
# Takes a dataset and multiplies the population column
# with the GDP per capita column.
calcGDP <- function(dat) {
gdp <- dat$pop * dat$gdpPercap
return(gdp)
}
We define calcGDP() by assigning it to the output of
function. The list of argument names are contained within
parentheses. Next, the body of the function -- the statements executed
when you call the function – is contained within curly braces
({}).
We’ve indented the statements in the body by two spaces. This makes the code easier to read but does not affect how it operates.
When we call the function, the values we pass to it are assigned to the arguments, which become variables inside the body of the function.
Inside the function, we use the return() function to
send back the result. This return() function is optional: R
will automatically return the results of whatever command is executed on
the last line of the function.
R
calcGDP(head(gapminder))
OUTPUT
[1] 6567086330 7585448670 8758855797 9648014150 9678553274 11697659231That’s not very informative. Let’s add some more arguments so we can extract that per year and country.
R
# Takes a dataset and multiplies the population column
# with the GDP per capita column.
calcGDP <- function(dat, year=NULL, country=NULL) {
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
gdp <- dat$pop * dat$gdpPercap
new <- cbind(dat, gdp=gdp)
return(new)
}
If you’ve been writing these functions down into a separate R script
(a good idea!), you can load in the functions into our R session by
using the source() function:
R
source("functions/functions-lesson.R")
Ok, so there’s a lot going on in this function now. In plain English, the function now subsets the provided data by year if the year argument isn’t empty, then subsets the result by country if the country argument isn’t empty. Then it calculates the GDP for whatever subset emerges from the previous two steps. The function then adds the GDP as a new column to the subsetted data and returns this as the final result. You can see that the output is much more informative than a vector of numbers.
Let’s take a look at what happens when we specify the year:
R
head(calcGDP(gapminder, year=2007))
OUTPUT
country year pop continent lifeExp gdpPercap gdp
12 Afghanistan 2007 31889923 Asia 43.828 974.5803 31079291949
24 Albania 2007 3600523 Europe 76.423 5937.0295 21376411360
36 Algeria 2007 33333216 Africa 72.301 6223.3675 207444851958
48 Angola 2007 12420476 Africa 42.731 4797.2313 59583895818
60 Argentina 2007 40301927 Americas 75.320 12779.3796 515033625357
72 Australia 2007 20434176 Oceania 81.235 34435.3674 703658358894Or for a specific country:
R
calcGDP(gapminder, country="Australia")
OUTPUT
country year pop continent lifeExp gdpPercap gdp
61 Australia 1952 8691212 Oceania 69.120 10039.60 87256254102
62 Australia 1957 9712569 Oceania 70.330 10949.65 106349227169
63 Australia 1962 10794968 Oceania 70.930 12217.23 131884573002
64 Australia 1967 11872264 Oceania 71.100 14526.12 172457986742
65 Australia 1972 13177000 Oceania 71.930 16788.63 221223770658
66 Australia 1977 14074100 Oceania 73.490 18334.20 258037329175
67 Australia 1982 15184200 Oceania 74.740 19477.01 295742804309
68 Australia 1987 16257249 Oceania 76.320 21888.89 355853119294
69 Australia 1992 17481977 Oceania 77.560 23424.77 409511234952
70 Australia 1997 18565243 Oceania 78.830 26997.94 501223252921
71 Australia 2002 19546792 Oceania 80.370 30687.75 599847158654
72 Australia 2007 20434176 Oceania 81.235 34435.37 703658358894Or both:
R
calcGDP(gapminder, year=2007, country="Australia")
OUTPUT
country year pop continent lifeExp gdpPercap gdp
72 Australia 2007 20434176 Oceania 81.235 34435.37 703658358894Let’s walk through the body of the function:
Here we’ve added two arguments, year, and
country. We’ve set default arguments for both as
NULL using the = operator in the function
definition. This means that those arguments will take on those values
unless the user specifies otherwise.
R
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
Here, we check whether each additional argument is set to
null, and whenever they’re not null overwrite
the dataset stored in dat with a subset given by the
non-null argument.
Building these conditionals into the function makes it more flexible for later. Now, we can use it to calculate the GDP for:
- The whole dataset;
- A single year;
- A single country;
- A single combination of year and country.
By using %in% instead, we can also give multiple years
or countries to those arguments.
Tip: Pass by value
Functions in R almost always make copies of the data to operate on
inside of a function body. When we modify dat inside the
function we are modifying the copy of the gapminder dataset stored in
dat, not the original variable we gave as the first
argument.
This is called “pass-by-value” and it makes writing code much safer: you can always be sure that whatever changes you make within the body of the function, stay inside the body of the function.
Tip: Function scope
Another important concept is scoping: any variables (or functions!)
you create or modify inside the body of a function only exist for the
lifetime of the function’s execution. When we call
calcGDP(), the variables dat, gdp
and new only exist inside the body of the function. Even if
we have variables of the same name in our interactive R session, they
are not modified in any way when executing a function.
Finally, we calculated the GDP on our new subset, and created a new data frame with that column added. This means when we call the function later we can see the context for the returned GDP values, which is much better than in our first attempt where we got a vector of numbers.
Challenge 4
Test out your GDP function by calculating the GDP for New Zealand in 1987. How does this differ from New Zealand’s GDP in 1952?
R
calcGDP(gapminder, year = c(1952, 1987), country = "New Zealand")
GDP for New Zealand in 1987: 65050008703
GDP for New Zealand in 1952: 21058193787
Challenge 5
The paste() function can be used to combine text
together, e.g:
R
best_practice <- c("Write", "programs", "for", "people", "not", "computers")
paste(best_practice, collapse=" ")
OUTPUT
[1] "Write programs for people not computers"Write a function called fence() that takes two vectors
as arguments, called text and wrapper, and
prints out the text wrapped with the wrapper:
R
fence(text=best_practice, wrapper="***")
Note: the paste() function has an argument
called sep, which specifies the separator between text. The
default is a space: ” “. The default for paste0() is no
space”“.
Write a function called fence() that takes two vectors
as arguments, called text and wrapper, and
prints out the text wrapped with the wrapper:
R
fence <- function(text, wrapper){
text <- c(wrapper, text, wrapper)
result <- paste(text, collapse = " ")
return(result)
}
best_practice <- c("Write", "programs", "for", "people", "not", "computers")
fence(text=best_practice, wrapper="***")
OUTPUT
[1] "*** Write programs for people not computers ***"Tip
R has some unique aspects that can be exploited when performing more complicated operations. We will not be writing anything that requires knowledge of these more advanced concepts. In the future when you are comfortable writing functions in R, you can learn more by reading the R Language Manual or this chapter from Advanced R Programming by Hadley Wickham.
Tip: Testing and documenting
It’s important to both test functions and document them: Documentation helps you, and others, understand what the purpose of your function is, and how to use it, and its important to make sure that your function actually does what you think.
When you first start out, your workflow will probably look a lot like this:
- Write a function
- Comment parts of the function to document its behaviour
- Load in the source file
- Experiment with it in the console to make sure it behaves as you expect
- Make any necessary bug fixes
- Rinse and repeat.
Formal documentation for functions, written in separate
.Rd files, gets turned into the documentation you see in
help files. The roxygen2
package allows R coders to write documentation alongside the function
code and then process it into the appropriate .Rd files.
You will want to switch to this more formal method of writing
documentation when you start writing more complicated R projects. In
fact, packages are, in essence, bundles of functions with this formal
documentation. Loading your own functions through
source("functions.R") is equivalent to loading someone
else’s functions (or your own one day!) through
library("package").
Formal automated tests can be written using the testthat package.
- Use
functionto define a new function in R. - Use parameters to pass values into functions.
- Use
stopifnot()to flexibly check function arguments in R. - Load functions into programs using
source().
Content from Writing Data
Last updated on 2025-07-01 | Edit this page
Estimated time: 20 minutes
Overview
Questions
- How can I save plots and data created in R?
Objectives
- To be able to write out plots and data from R.
Saving plots
You have already seen how to save the most recent plot you create in
ggplot2, using the command ggsave. As a
refresher:
R
ggsave("My_most_recent_plot.pdf")
You can save a plot from within RStudio using the ‘Export’ button in the ‘Plot’ window. This will give you the option of saving as a .pdf or as .png, .jpg or other image formats.
Sometimes you will want to save plots without creating them in the ‘Plot’ window first. Perhaps you want to make a pdf document with multiple pages: each one a different plot, for example. Or perhaps you’re looping through multiple subsets of a file, plotting data from each subset, and you want to save each plot, but obviously can’t stop the loop to click ‘Export’ for each one.
In this case you can use a more flexible approach. The function
pdf creates a new pdf device. You can control the size and
resolution using the arguments to this function.
R
pdf("Life_Exp_vs_time.pdf", width=12, height=4)
ggplot(data=gapminder, aes(x=year, y=lifeExp, colour=country)) +
geom_line() +
theme(legend.position = "none")
# You then have to make sure to turn off the pdf device!
dev.off()
Open up this document and have a look.
Challenge 1
Rewrite your ‘pdf’ command to print a second page in the pdf, showing
a facet plot (hint: use facet_grid) of the same data with
one panel per continent.
R
pdf("Life_Exp_vs_time.pdf", width = 12, height = 4)
p <- ggplot(data = gapminder, aes(x = year, y = lifeExp, colour = country)) +
geom_line() +
theme(legend.position = "none")
p
p + facet_grid(~continent)
dev.off()
The commands jpeg, png etc. are used
similarly to produce documents in different formats.
Writing data
At some point, you’ll also want to write out data from R.
We can use the write.table function for this, which is
very similar to read.table from before.
Let’s create a data-cleaning script, for this analysis, we only want to focus on the gapminder data for Australia:
R
aust_subset <- gapminder[gapminder$country == "Australia",]
write.table(aust_subset,
file="cleaned-data/gapminder-aus.csv",
sep=","
)
Let’s switch back to the shell to take a look at the data to make sure it looks OK:
OUTPUT
"country","year","pop","continent","lifeExp","gdpPercap"
"61","Australia",1952,8691212,"Oceania",69.12,10039.59564
"62","Australia",1957,9712569,"Oceania",70.33,10949.64959
"63","Australia",1962,10794968,"Oceania",70.93,12217.22686
"64","Australia",1967,11872264,"Oceania",71.1,14526.12465
"65","Australia",1972,13177000,"Oceania",71.93,16788.62948
"66","Australia",1977,14074100,"Oceania",73.49,18334.19751
"67","Australia",1982,15184200,"Oceania",74.74,19477.00928
"68","Australia",1987,16257249,"Oceania",76.32,21888.88903
"69","Australia",1992,17481977,"Oceania",77.56,23424.76683Hmm, that’s not quite what we wanted. Where did all these quotation marks come from? Also the row numbers are meaningless.
Let’s look at the help file to work out how to change this behaviour.
R
?write.table
By default R will wrap character vectors with quotation marks when writing out to file. It will also write out the row and column names.
Let’s fix this:
R
write.table(
gapminder[gapminder$country == "Australia",],
file="cleaned-data/gapminder-aus.csv",
sep=",", quote=FALSE, row.names=FALSE
)
Now lets look at the data again using our shell skills:
OUTPUT
country,year,pop,continent,lifeExp,gdpPercap
Australia,1952,8691212,Oceania,69.12,10039.59564
Australia,1957,9712569,Oceania,70.33,10949.64959
Australia,1962,10794968,Oceania,70.93,12217.22686
Australia,1967,11872264,Oceania,71.1,14526.12465
Australia,1972,13177000,Oceania,71.93,16788.62948
Australia,1977,14074100,Oceania,73.49,18334.19751
Australia,1982,15184200,Oceania,74.74,19477.00928
Australia,1987,16257249,Oceania,76.32,21888.88903
Australia,1992,17481977,Oceania,77.56,23424.76683That looks better!
Challenge 2
Write a data-cleaning script file that subsets the gapminder data to include only data points collected since 1990.
Use this script to write out the new subset to a file in the
cleaned-data/ directory.
R
write.table(
gapminder[gapminder$year > 1990, ],
file = "cleaned-data/gapminder-after1990.csv",
sep = ",", quote = FALSE, row.names = FALSE
)
- Save plots from RStudio using the ‘Export’ button.
- Use
write.tableto save tabular data.
Content from Data Frame Manipulation with dplyr
Last updated on 2025-07-01 | Edit this page
Estimated time: 55 minutes
Overview
Questions
- How can I manipulate data frames without repeating myself?
Objectives
- To be able to use the six main data frame manipulation ‘verbs’ with
pipes in
dplyr. - To understand how
group_by()andsummarize()can be combined to summarize datasets. - Be able to analyze a subset of data using logical filtering.
Manipulation of data frames means many things to many researchers: we often select certain observations (rows) or variables (columns), we often group the data by a certain variable(s), or we even calculate summary statistics. We can do these operations using the normal base R operations:
R
mean(gapminder$gdpPercap[gapminder$continent == "Africa"])
OUTPUT
[1] 2193.755R
mean(gapminder$gdpPercap[gapminder$continent == "Americas"])
OUTPUT
[1] 7136.11R
mean(gapminder$gdpPercap[gapminder$continent == "Asia"])
OUTPUT
[1] 7902.15But this isn’t very nice because there is a fair bit of repetition. Repeating yourself will cost you time, both now and later, and potentially introduce some nasty bugs.
The dplyr package
Luckily, the dplyr
package provides a number of very useful functions for manipulating data
frames in a way that will reduce the above repetition, reduce the
probability of making errors, and probably even save you some typing. As
an added bonus, you might even find the dplyr grammar
easier to read.
Tip: Tidyverse
dplyr package belongs to a broader family of opinionated
R packages designed for data science called the “Tidyverse”. These
packages are specifically designed to work harmoniously together. Some
of these packages will be covered along this course, but you can find
more complete information here: https://www.tidyverse.org/.
Here we’re going to cover 5 of the most commonly used functions as
well as using pipes (%>%) to combine them.
select()filter()group_by()summarize()mutate()
If you have have not installed this package earlier, please do so:
R
install.packages('dplyr')
Now let’s load the package:
R
library("dplyr")
Using select()
If, for example, we wanted to move forward with only a few of the
variables in our data frame we could use the select()
function. This will keep only the variables you select.
R
year_country_gdp <- select(gapminder, year, country, gdpPercap)
 If we want to remove one column only from the
If we want to remove one column only from the gapminder
data, for example, removing the continent column.
R
smaller_gapminder_data <- select(gapminder, -continent)
If we open up year_country_gdp we’ll see that it only
contains the year, country and gdpPercap. Above we used ‘normal’
grammar, but the strengths of dplyr lie in combining
several functions using pipes. Since the pipes grammar is unlike
anything we’ve seen in R before, let’s repeat what we’ve done above
using pipes.
R
year_country_gdp <- gapminder %>% select(year, country, gdpPercap)
To help you understand why we wrote that in that way, let’s walk
through it step by step. First we summon the gapminder data frame and
pass it on, using the pipe symbol %>%, to the next step,
which is the select() function. In this case we don’t
specify which data object we use in the select() function
since in gets that from the previous pipe. Fun Fact:
There is a good chance you have encountered pipes before in the shell.
In R, a pipe symbol is %>% while in the shell it is
| but the concept is the same!
Tip: Renaming data frame columns in dplyr
In Chapter 4 we covered how you can rename columns with base R by
assigning a value to the output of the names() function.
Just like select, this is a bit cumbersome, but thankfully dplyr has a
rename() function.
Within a pipeline, the syntax is
rename(new_name = old_name). For example, we may want to
rename the gdpPercap column name from our select()
statement above.
R
tidy_gdp <- year_country_gdp %>% rename(gdp_per_capita = gdpPercap)
head(tidy_gdp)
OUTPUT
year country gdp_per_capita
1 1952 Afghanistan 779.4453
2 1957 Afghanistan 820.8530
3 1962 Afghanistan 853.1007
4 1967 Afghanistan 836.1971
5 1972 Afghanistan 739.9811
6 1977 Afghanistan 786.1134Using filter()
If we now want to move forward with the above, but only with European
countries, we can combine select and
filter
R
year_country_gdp_euro <- gapminder %>%
filter(continent == "Europe") %>%
select(year, country, gdpPercap)
If we now want to show life expectancy of European countries but only for a specific year (e.g., 2007), we can do as below.
R
europe_lifeExp_2007 <- gapminder %>%
filter(continent == "Europe", year == 2007) %>%
select(country, lifeExp)
Challenge 1
Write a single command (which can span multiple lines and includes
pipes) that will produce a data frame that has the African values for
lifeExp, country and year, but
not for other Continents. How many rows does your data frame have and
why?
R
year_country_lifeExp_Africa <- gapminder %>%
filter(continent == "Africa") %>%
select(year, country, lifeExp)
As with last time, first we pass the gapminder data frame to the
filter() function, then we pass the filtered version of the
gapminder data frame to the select() function.
Note: The order of operations is very important in this
case. If we used ‘select’ first, filter would not be able to find the
variable continent since we would have removed it in the previous
step.
Using group_by()
Now, we were supposed to be reducing the error prone repetitiveness
of what can be done with base R, but up to now we haven’t done that
since we would have to repeat the above for each continent. Instead of
filter(), which will only pass observations that meet your
criteria (in the above: continent=="Europe"), we can use
group_by(), which will essentially use every unique
criteria that you could have used in filter.
R
str(gapminder)
OUTPUT
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...R
str(gapminder %>% group_by(continent))
OUTPUT
gropd_df [1,704 × 6] (S3: grouped_df/tbl_df/tbl/data.frame)
$ country : chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int [1:1704] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num [1:1704] 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr [1:1704] "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num [1:1704] 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num [1:1704] 779 821 853 836 740 ...
- attr(*, "groups")= tibble [5 × 2] (S3: tbl_df/tbl/data.frame)
..$ continent: chr [1:5] "Africa" "Americas" "Asia" "Europe" ...
..$ .rows : list<int> [1:5]
.. ..$ : int [1:624] 25 26 27 28 29 30 31 32 33 34 ...
.. ..$ : int [1:300] 49 50 51 52 53 54 55 56 57 58 ...
.. ..$ : int [1:396] 1 2 3 4 5 6 7 8 9 10 ...
.. ..$ : int [1:360] 13 14 15 16 17 18 19 20 21 22 ...
.. ..$ : int [1:24] 61 62 63 64 65 66 67 68 69 70 ...
.. ..@ ptype: int(0)
..- attr(*, ".drop")= logi TRUEYou will notice that the structure of the data frame where we used
group_by() (grouped_df) is not the same as the
original gapminder (data.frame). A
grouped_df can be thought of as a list where
each item in the listis a data.frame which
contains only the rows that correspond to the a particular value
continent (at least in the example above).
Using summarize()
The above was a bit on the uneventful side but
group_by() is much more exciting in conjunction with
summarize(). This will allow us to create new variable(s)
by using functions that repeat for each of the continent-specific data
frames. That is to say, using the group_by() function, we
split our original data frame into multiple pieces, then we can run
functions (e.g. mean() or sd()) within
summarize().
R
gdp_bycontinents <- gapminder %>%
group_by(continent) %>%
summarize(mean_gdpPercap = mean(gdpPercap))
R
continent mean_gdpPercap
<fctr> <dbl>
1 Africa 2193.755
2 Americas 7136.110
3 Asia 7902.150
4 Europe 14469.476
5 Oceania 18621.609That allowed us to calculate the mean gdpPercap for each continent, but it gets even better.
Challenge 2
Calculate the average life expectancy per country. Which has the longest average life expectancy and which has the shortest average life expectancy?
R
lifeExp_bycountry <- gapminder %>%
group_by(country) %>%
summarize(mean_lifeExp = mean(lifeExp))
lifeExp_bycountry %>%
filter(mean_lifeExp == min(mean_lifeExp) | mean_lifeExp == max(mean_lifeExp))
OUTPUT
# A tibble: 2 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5
2 Sierra Leone 36.8Another way to do this is to use the dplyr function
arrange(), which arranges the rows in a data frame
according to the order of one or more variables from the data frame. It
has similar syntax to other functions from the dplyr
package. You can use desc() inside arrange()
to sort in descending order.
R
lifeExp_bycountry %>%
arrange(mean_lifeExp) %>%
head(1)
OUTPUT
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Sierra Leone 36.8R
lifeExp_bycountry %>%
arrange(desc(mean_lifeExp)) %>%
head(1)
OUTPUT
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5Alphabetical order works too
R
lifeExp_bycountry %>%
arrange(desc(country)) %>%
head(1)
OUTPUT
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Zimbabwe 52.7The function group_by() allows us to group by multiple
variables. Let’s group by year and
continent.
R
gdp_bycontinents_byyear <- gapminder %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap))
OUTPUT
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.That is already quite powerful, but it gets even better! You’re not
limited to defining 1 new variable in summarize().
R
gdp_pop_bycontinents_byyear <- gapminder %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop))
OUTPUT
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.count() and n()
A very common operation is to count the number of observations for
each group. The dplyr package comes with two related
functions that help with this.
For instance, if we wanted to check the number of countries included
in the dataset for the year 2002, we can use the count()
function. It takes the name of one or more columns that contain the
groups we are interested in, and we can optionally sort the results in
descending order by adding sort=TRUE:
R
gapminder %>%
filter(year == 2002) %>%
count(continent, sort = TRUE)
OUTPUT
continent n
1 Africa 52
2 Asia 33
3 Europe 30
4 Americas 25
5 Oceania 2If we need to use the number of observations in calculations, the
n() function is useful. It will return the total number of
observations in the current group rather than counting the number of
observations in each group within a specific column. For instance, if we
wanted to get the standard error of the life expectency per
continent:
R
gapminder %>%
group_by(continent) %>%
summarize(se_le = sd(lifeExp)/sqrt(n()))
OUTPUT
# A tibble: 5 × 2
continent se_le
<chr> <dbl>
1 Africa 0.366
2 Americas 0.540
3 Asia 0.596
4 Europe 0.286
5 Oceania 0.775You can also chain together several summary operations; in this case
calculating the minimum, maximum,
mean and se of each continent’s per-country
life-expectancy:
R
gapminder %>%
group_by(continent) %>%
summarize(
mean_le = mean(lifeExp),
min_le = min(lifeExp),
max_le = max(lifeExp),
se_le = sd(lifeExp)/sqrt(n()))
OUTPUT
# A tibble: 5 × 5
continent mean_le min_le max_le se_le
<chr> <dbl> <dbl> <dbl> <dbl>
1 Africa 48.9 23.6 76.4 0.366
2 Americas 64.7 37.6 80.7 0.540
3 Asia 60.1 28.8 82.6 0.596
4 Europe 71.9 43.6 81.8 0.286
5 Oceania 74.3 69.1 81.2 0.775Using mutate()
We can also create new variables prior to (or even after) summarizing
information using mutate().
R
gdp_pop_bycontinents_byyear <- gapminder %>%
mutate(gdp_billion = gdpPercap*pop/10^9) %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
OUTPUT
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.Connect mutate with logical filtering: ifelse
When creating new variables, we can hook this with a logical
condition. A simple combination of mutate() and
ifelse() facilitates filtering right where it is needed: in
the moment of creating something new. This easy-to-read statement is a
fast and powerful way of discarding certain data (even though the
overall dimension of the data frame will not change) or for updating
values depending on this given condition.
R
## keeping all data but "filtering" after a certain condition
# calculate GDP only for people with a life expectation above 25
gdp_pop_bycontinents_byyear_above25 <- gapminder %>%
mutate(gdp_billion = ifelse(lifeExp > 25, gdpPercap * pop / 10^9, NA)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
OUTPUT
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.R
## updating only if certain condition is fullfilled
# for life expectations above 40 years, the gpd to be expected in the future is scaled
gdp_future_bycontinents_byyear_high_lifeExp <- gapminder %>%
mutate(gdp_futureExpectation = ifelse(lifeExp > 40, gdpPercap * 1.5, gdpPercap)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
mean_gdpPercap_expected = mean(gdp_futureExpectation))
OUTPUT
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.Combining dplyr and ggplot2
First install and load ggplot2:
R
install.packages('ggplot2')
R
library("ggplot2")
In the plotting lesson we looked at how to make a multi-panel figure
by adding a layer of facet panels using ggplot2. Here is
the code we used (with some extra comments):
R
# Filter countries located in the Americas
americas <- gapminder[gapminder$continent == "Americas", ]
# Make the plot
ggplot(data = americas, mapping = aes(x = year, y = lifeExp)) +
geom_line() +
facet_wrap( ~ country) +
theme(axis.text.x = element_text(angle = 45))
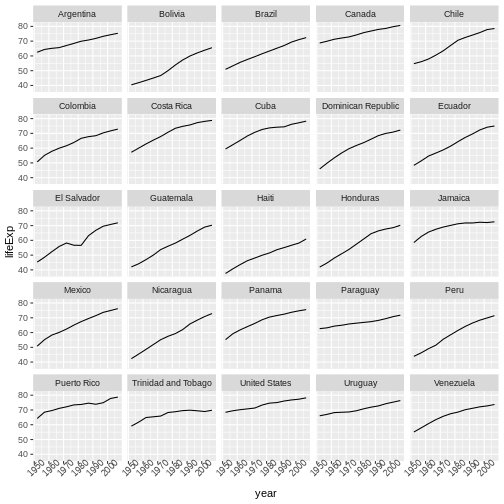
This code makes the right plot but it also creates an intermediate
variable (americas) that we might not have any other uses
for. Just as we used %>% to pipe data along a chain of
dplyr functions we can use it to pass data to
ggplot(). Because %>% replaces the first
argument in a function we don’t need to specify the data =
argument in the ggplot() function. By combining
dplyr and ggplot2 functions we can make the
same figure without creating any new variables or modifying the
data.
R
gapminder %>%
# Filter countries located in the Americas
filter(continent == "Americas") %>%
# Make the plot
ggplot(mapping = aes(x = year, y = lifeExp)) +
geom_line() +
facet_wrap( ~ country) +
theme(axis.text.x = element_text(angle = 45))
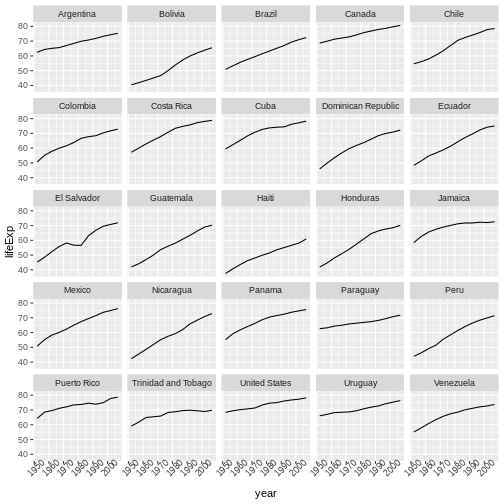
More examples of using the function mutate() and the
ggplot2 package.
R
gapminder %>%
# extract first letter of country name into new column
mutate(startsWith = substr(country, 1, 1)) %>%
# only keep countries starting with A or Z
filter(startsWith %in% c("A", "Z")) %>%
# plot lifeExp into facets
ggplot(aes(x = year, y = lifeExp, colour = continent)) +
geom_line() +
facet_wrap(vars(country)) +
theme_minimal()
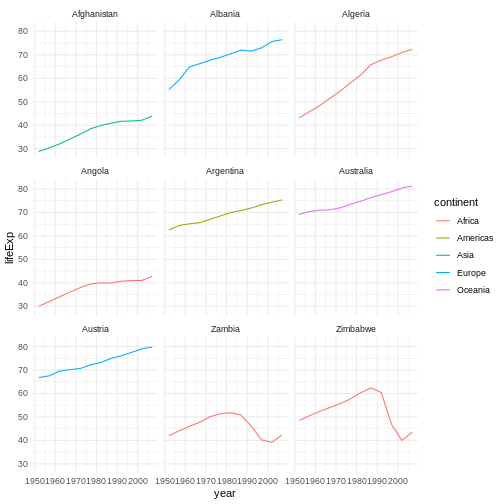
Advanced Challenge
Calculate the average life expectancy in 2002 of 2 randomly selected
countries for each continent. Then arrange the continent names in
reverse order. Hint: Use the dplyr
functions arrange() and sample_n(), they have
similar syntax to other dplyr functions.
R
lifeExp_2countries_bycontinents <- gapminder %>%
filter(year==2002) %>%
group_by(continent) %>%
sample_n(2) %>%
summarize(mean_lifeExp=mean(lifeExp)) %>%
arrange(desc(mean_lifeExp))
Other great resources
- R for Data Science (online book)
- Data Wrangling Cheat sheet (pdf file)
- Introduction to dplyr (online documentation)
- Data wrangling with R and RStudio (online video)
- Tidyverse Skills for Data Science (online book)
- Use the
dplyrpackage to manipulate data frames. - Use
select()to choose variables from a data frame. - Use
filter()to choose data based on values. - Use
group_by()andsummarize()to work with subsets of data. - Use
mutate()to create new variables.
Content from Data Frame Manipulation with tidyr
Last updated on 2025-07-01 | Edit this page
Estimated time: 45 minutes
Overview
Questions
- How can I change the layout of a data frame?
Objectives
- To understand the concepts of ‘longer’ and ‘wider’ data frame
formats and be able to convert between them with
tidyr.
Researchers often want to reshape their data frames from ‘wide’ to ‘longer’ layouts, or vice-versa. The ‘long’ layout or format is where:
- each column is a variable
- each row is an observation
In the purely ‘long’ (or ‘longest’) format, you usually have 1 column for the observed variable and the other columns are ID variables.
For the ‘wide’ format each row is often a site/subject/patient and
you have multiple observation variables containing the same type of
data. These can be either repeated observations over time, or
observation of multiple variables (or a mix of both). You may find data
input may be simpler or some other applications may prefer the ‘wide’
format. However, many of R‘s functions have been designed
assuming you have ’longer’ formatted data. This tutorial will help you
efficiently transform your data shape regardless of original format.
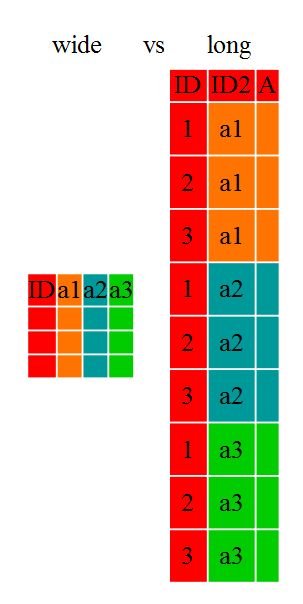
Long and wide data frame layouts mainly affect readability. For humans, the wide format is often more intuitive since we can often see more of the data on the screen due to its shape. However, the long format is more machine readable and is closer to the formatting of databases. The ID variables in our data frames are similar to the fields in a database and observed variables are like the database values.
Getting started
First install the packages if you haven’t already done so (you probably installed dplyr in the previous lesson):
R
#install.packages("tidyr")
#install.packages("dplyr")
Load the packages
R
library("tidyr")
library("dplyr")
First, lets look at the structure of our original gapminder data frame:
R
str(gapminder)
OUTPUT
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...Challenge 1
Is gapminder a purely long, purely wide, or some intermediate format?
The original gapminder data.frame is in an intermediate format. It is
not purely long since it had multiple observation variables
(pop,lifeExp,gdpPercap).
Sometimes, as with the gapminder dataset, we have multiple types of
observed data. It is somewhere in between the purely ‘long’ and ‘wide’
data formats. We have 3 “ID variables” (continent,
country, year) and 3 “Observation variables”
(pop,lifeExp,gdpPercap). This
intermediate format can be preferred despite not having ALL observations
in 1 column given that all 3 observation variables have different units.
There are few operations that would need us to make this data frame any
longer (i.e. 4 ID variables and 1 Observation variable).
While using many of the functions in R, which are often vector based,
you usually do not want to do mathematical operations on values with
different units. For example, using the purely long format, a single
mean for all of the values of population, life expectancy, and GDP would
not be meaningful since it would return the mean of values with 3
incompatible units. The solution is that we first manipulate the data
either by grouping (see the lesson on dplyr), or we change
the structure of the data frame. Note: Some plotting
functions in R actually work better in the wide format data.
From wide to long format with pivot_longer()
Until now, we’ve been using the nicely formatted original gapminder dataset, but ‘real’ data (i.e. our own research data) will never be so well organized. Here let’s start with the wide formatted version of the gapminder dataset.
Download the wide version of the gapminder data from this link to a csv file and save it in your data folder.
We’ll load the data file and look at it. Note: we don’t want our
continent and country columns to be factors, so we use the
stringsAsFactors argument for read.csv() to disable
that.
R
gap_wide <- read.csv("data/gapminder_wide.csv", stringsAsFactors = FALSE)
str(gap_wide)
OUTPUT
'data.frame': 142 obs. of 38 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : int 31287142 10866106 7026113 1630347 12251209 7021078 15929988 4048013 8835739 614382 ...
$ pop_2007 : int 33333216 12420476 8078314 1639131 14326203 8390505 17696293 4369038 10238807 710960 ...
To change this very wide data frame layout back to our nice,
intermediate (or longer) layout, we will use one of the two available
pivot functions from the tidyr package. To
convert from wide to a longer format, we will use the
pivot_longer() function. pivot_longer() makes
datasets longer by increasing the number of rows and decreasing the
number of columns, or ‘lengthening’ your observation variables into a
single variable.
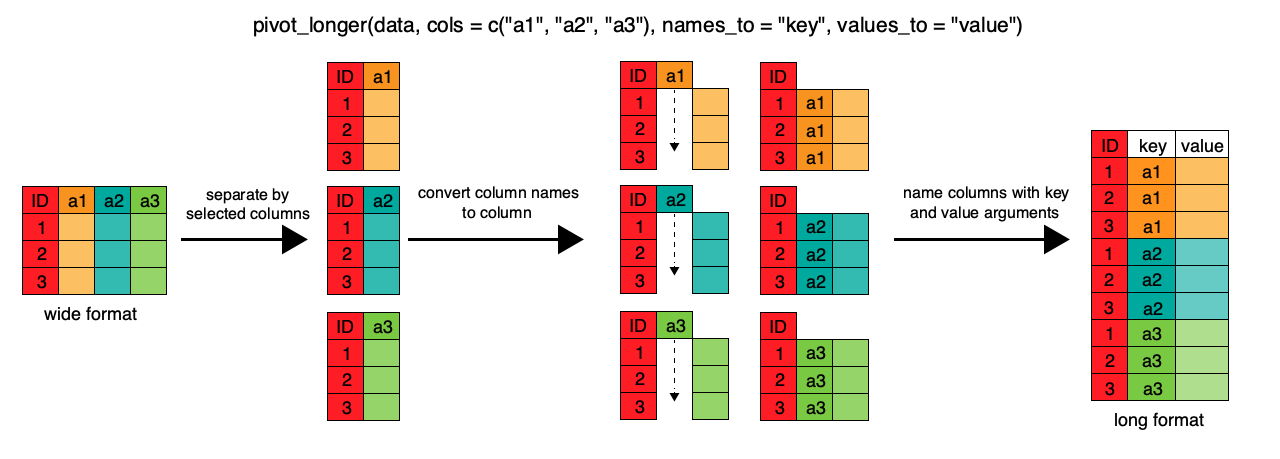
R
gap_long <- gap_wide %>%
pivot_longer(
cols = c(starts_with('pop'), starts_with('lifeExp'), starts_with('gdpPercap')),
names_to = "obstype_year", values_to = "obs_values"
)
str(gap_long)
OUTPUT
tibble [5,112 × 4] (S3: tbl_df/tbl/data.frame)
$ continent : chr [1:5112] "Africa" "Africa" "Africa" "Africa" ...
$ country : chr [1:5112] "Algeria" "Algeria" "Algeria" "Algeria" ...
$ obstype_year: chr [1:5112] "pop_1952" "pop_1957" "pop_1962" "pop_1967" ...
$ obs_values : num [1:5112] 9279525 10270856 11000948 12760499 14760787 ...Here we have used piping syntax which is similar to what we were doing in the previous lesson with dplyr. In fact, these are compatible and you can use a mix of tidyr and dplyr functions by piping them together.
We first provide to pivot_longer() a vector of column
names that will be pivoted into longer format. We could type out all the
observation variables, but as in the select() function (see
dplyr lesson), we can use the starts_with()
argument to select all variables that start with the desired character
string. pivot_longer() also allows the alternative syntax
of using the - symbol to identify which variables are not
to be pivoted (i.e. ID variables).
The next arguments to pivot_longer() are
names_to for naming the column that will contain the new ID
variable (obstype_year) and values_to for
naming the new amalgamated observation variable
(obs_value). We supply these new column names as
strings.
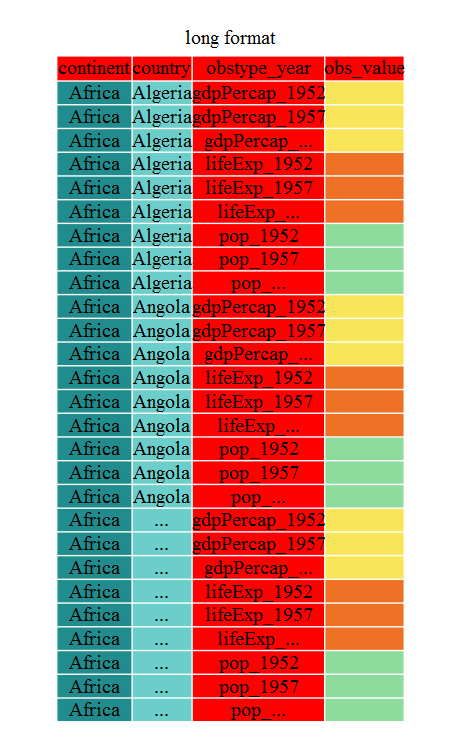
R
gap_long <- gap_wide %>%
pivot_longer(
cols = c(-continent, -country),
names_to = "obstype_year", values_to = "obs_values"
)
str(gap_long)
OUTPUT
tibble [5,112 × 4] (S3: tbl_df/tbl/data.frame)
$ continent : chr [1:5112] "Africa" "Africa" "Africa" "Africa" ...
$ country : chr [1:5112] "Algeria" "Algeria" "Algeria" "Algeria" ...
$ obstype_year: chr [1:5112] "gdpPercap_1952" "gdpPercap_1957" "gdpPercap_1962" "gdpPercap_1967" ...
$ obs_values : num [1:5112] 2449 3014 2551 3247 4183 ...That may seem trivial with this particular data frame, but sometimes you have 1 ID variable and 40 observation variables with irregular variable names. The flexibility is a huge time saver!
Now obstype_year actually contains 2 pieces of
information, the observation type
(pop,lifeExp, or gdpPercap) and
the year. We can use the separate() function
to split the character strings into multiple variables
R
gap_long <- gap_long %>% separate(obstype_year, into = c('obs_type', 'year'), sep = "_")
gap_long$year <- as.integer(gap_long$year)
Challenge 2
Using gap_long, calculate the mean life expectancy,
population, and gdpPercap for each continent. Hint: use
the group_by() and summarize() functions we
learned in the dplyr lesson
R
gap_long %>% group_by(continent, obs_type) %>%
summarize(means=mean(obs_values))
OUTPUT
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.OUTPUT
# A tibble: 15 × 3
# Groups: continent [5]
continent obs_type means
<chr> <chr> <dbl>
1 Africa gdpPercap 2194.
2 Africa lifeExp 48.9
3 Africa pop 9916003.
4 Americas gdpPercap 7136.
5 Americas lifeExp 64.7
6 Americas pop 24504795.
7 Asia gdpPercap 7902.
8 Asia lifeExp 60.1
9 Asia pop 77038722.
10 Europe gdpPercap 14469.
11 Europe lifeExp 71.9
12 Europe pop 17169765.
13 Oceania gdpPercap 18622.
14 Oceania lifeExp 74.3
15 Oceania pop 8874672. From long to intermediate format with pivot_wider()
It is always good to check work. So, let’s use the second
pivot function, pivot_wider(), to ‘widen’ our
observation variables back out. pivot_wider() is the
opposite of pivot_longer(), making a dataset wider by
increasing the number of columns and decreasing the number of rows. We
can use pivot_wider() to pivot or reshape our
gap_long to the original intermediate format or the widest
format. Let’s start with the intermediate format.
The pivot_wider() function takes names_from
and values_from arguments.
To names_from we supply the column name whose contents
will be pivoted into new output columns in the widened data frame. The
corresponding values will be added from the column named in the
values_from argument.
R
gap_normal <- gap_long %>%
pivot_wider(names_from = obs_type, values_from = obs_values)
dim(gap_normal)
OUTPUT
[1] 1704 6R
dim(gapminder)
OUTPUT
[1] 1704 6R
names(gap_normal)
OUTPUT
[1] "continent" "country" "year" "gdpPercap" "lifeExp" "pop" R
names(gapminder)
OUTPUT
[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"Now we’ve got an intermediate data frame gap_normal with
the same dimensions as the original gapminder, but the
order of the variables is different. Let’s fix that before checking if
they are all.equal().
R
gap_normal <- gap_normal[, names(gapminder)]
all.equal(gap_normal, gapminder)
OUTPUT
[1] "Attributes: < Component \"class\": Lengths (3, 1) differ (string compare on first 1) >"
[2] "Attributes: < Component \"class\": 1 string mismatch >"
[3] "Component \"country\": 1704 string mismatches"
[4] "Component \"pop\": Mean relative difference: 1.634504"
[5] "Component \"continent\": 1212 string mismatches"
[6] "Component \"lifeExp\": Mean relative difference: 0.203822"
[7] "Component \"gdpPercap\": Mean relative difference: 1.162302" R
head(gap_normal)
OUTPUT
# A tibble: 6 × 6
country year pop continent lifeExp gdpPercap
<chr> <int> <dbl> <chr> <dbl> <dbl>
1 Algeria 1952 9279525 Africa 43.1 2449.
2 Algeria 1957 10270856 Africa 45.7 3014.
3 Algeria 1962 11000948 Africa 48.3 2551.
4 Algeria 1967 12760499 Africa 51.4 3247.
5 Algeria 1972 14760787 Africa 54.5 4183.
6 Algeria 1977 17152804 Africa 58.0 4910.R
head(gapminder)
OUTPUT
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134We’re almost there, the original was sorted by country,
then year.
R
gap_normal <- gap_normal %>% arrange(country, year)
all.equal(gap_normal, gapminder)
OUTPUT
[1] "Attributes: < Component \"class\": Lengths (3, 1) differ (string compare on first 1) >"
[2] "Attributes: < Component \"class\": 1 string mismatch >" That’s great! We’ve gone from the longest format back to the intermediate and we didn’t introduce any errors in our code.
Now let’s convert the long all the way back to the wide. In the wide
format, we will keep country and continent as ID variables and pivot the
observations across the 3 metrics
(pop,lifeExp,gdpPercap) and time
(year). First we need to create appropriate labels for all
our new variables (time*metric combinations) and we also need to unify
our ID variables to simplify the process of defining
gap_wide.
R
gap_temp <- gap_long %>% unite(var_ID, continent, country, sep = "_")
str(gap_temp)
OUTPUT
tibble [5,112 × 4] (S3: tbl_df/tbl/data.frame)
$ var_ID : chr [1:5112] "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" ...
$ obs_type : chr [1:5112] "gdpPercap" "gdpPercap" "gdpPercap" "gdpPercap" ...
$ year : int [1:5112] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ obs_values: num [1:5112] 2449 3014 2551 3247 4183 ...R
gap_temp <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_")
str(gap_temp)
OUTPUT
tibble [5,112 × 3] (S3: tbl_df/tbl/data.frame)
$ ID_var : chr [1:5112] "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" ...
$ var_names : chr [1:5112] "gdpPercap_1952" "gdpPercap_1957" "gdpPercap_1962" "gdpPercap_1967" ...
$ obs_values: num [1:5112] 2449 3014 2551 3247 4183 ...Using unite() we now have a single ID variable which is
a combination of continent,country,and we have
defined variable names. We’re now ready to pipe in
pivot_wider()
R
gap_wide_new <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_") %>%
pivot_wider(names_from = var_names, values_from = obs_values)
str(gap_wide_new)
OUTPUT
tibble [142 × 37] (S3: tbl_df/tbl/data.frame)
$ ID_var : chr [1:142] "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ gdpPercap_1952: num [1:142] 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num [1:142] 3014 3828 960 918 617 ...
$ gdpPercap_1962: num [1:142] 2551 4269 949 984 723 ...
$ gdpPercap_1967: num [1:142] 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num [1:142] 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num [1:142] 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num [1:142] 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num [1:142] 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num [1:142] 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num [1:142] 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num [1:142] 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num [1:142] 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num [1:142] 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num [1:142] 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num [1:142] 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num [1:142] 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num [1:142] 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num [1:142] 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num [1:142] 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num [1:142] 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num [1:142] 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num [1:142] 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num [1:142] 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num [1:142] 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num [1:142] 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num [1:142] 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num [1:142] 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num [1:142] 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num [1:142] 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num [1:142] 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num [1:142] 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num [1:142] 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num [1:142] 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num [1:142] 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num [1:142] 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num [1:142] 33333216 12420476 8078314 1639131 14326203 ...Challenge 3
Take this 1 step further and create a
gap_ludicrously_wide format data by pivoting over
countries, year and the 3 metrics? Hint this new data
frame should only have 5 rows.
R
gap_ludicrously_wide <- gap_long %>%
unite(var_names, obs_type, year, country, sep = "_") %>%
pivot_wider(names_from = var_names, values_from = obs_values)
Now we have a great ‘wide’ format data frame, but the
ID_var could be more usable, let’s separate it into 2
variables with separate()
R
gap_wide_betterID <- separate(gap_wide_new, ID_var, c("continent", "country"), sep="_")
gap_wide_betterID <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_") %>%
pivot_wider(names_from = var_names, values_from = obs_values) %>%
separate(ID_var, c("continent","country"), sep = "_")
str(gap_wide_betterID)
OUTPUT
tibble [142 × 38] (S3: tbl_df/tbl/data.frame)
$ continent : chr [1:142] "Africa" "Africa" "Africa" "Africa" ...
$ country : chr [1:142] "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num [1:142] 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num [1:142] 3014 3828 960 918 617 ...
$ gdpPercap_1962: num [1:142] 2551 4269 949 984 723 ...
$ gdpPercap_1967: num [1:142] 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num [1:142] 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num [1:142] 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num [1:142] 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num [1:142] 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num [1:142] 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num [1:142] 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num [1:142] 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num [1:142] 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num [1:142] 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num [1:142] 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num [1:142] 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num [1:142] 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num [1:142] 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num [1:142] 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num [1:142] 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num [1:142] 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num [1:142] 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num [1:142] 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num [1:142] 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num [1:142] 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num [1:142] 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num [1:142] 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num [1:142] 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num [1:142] 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num [1:142] 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num [1:142] 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num [1:142] 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num [1:142] 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num [1:142] 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num [1:142] 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num [1:142] 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num [1:142] 33333216 12420476 8078314 1639131 14326203 ...R
all.equal(gap_wide, gap_wide_betterID)
OUTPUT
[1] "Attributes: < Component \"class\": Lengths (1, 3) differ (string compare on first 1) >"
[2] "Attributes: < Component \"class\": 1 string mismatch >" There and back again!
Other great resources
- R for Data Science (online book)
- Data Wrangling Cheat sheet (pdf file)
- Introduction to tidyr (online documentation)
- Data wrangling with R and RStudio (online video)
- Use the
tidyrpackage to change the layout of data frames. - Use
pivot_longer()to go from wide to longer layout. - Use
pivot_wider()to go from long to wider layout.
Content from Producing Reports With knitr
Last updated on 2025-07-01 | Edit this page
Estimated time: 75 minutes
Overview
Questions
- How can I integrate software and reports?
Objectives
- Understand the value of writing reproducible reports
- Learn how to recognise and compile the basic components of an R Markdown file
- Become familiar with R code chunks, and understand their purpose, structure and options
- Demonstrate the use of inline chunks for weaving R outputs into text blocks, for example when discussing the results of some calculations
- Be aware of alternative output formats to which an R Markdown file can be exported
Data analysis reports
Data analysts tend to write a lot of reports, describing their analyses and results, for their collaborators or to document their work for future reference.
Many new users begin by first writing a single R script containing all of their work, and then share the analysis by emailing the script and various graphs as attachments. But this can be cumbersome, requiring a lengthy discussion to explain which attachment was which result.
Writing formal reports with Word or LaTeX can simplify this process by incorporating both the analysis report and output graphs into a single document. But tweaking formatting to make figures look correct and fixing obnoxious page breaks can be tedious and lead to a lengthy “whack-a-mole” game of fixing new mistakes resulting from a single formatting change.
Creating a report as a web page (which is an html file) using R Markdown makes things easier. The report can be one long stream, so tall figures that wouldn’t ordinarily fit on one page can be kept at full size and easier to read, since the reader can simply keep scrolling. Additionally, the formatting of and R Markdown document is simple and easy to modify, allowing you to spend more time on your analyses instead of writing reports.
Literate programming
Ideally, such analysis reports are reproducible documents: If an error is discovered, or if some additional subjects are added to the data, you can just re-compile the report and get the new or corrected results rather than having to reconstruct figures, paste them into a Word document, and hand-edit various detailed results.
The key R package here is knitr. It allows you
to create a document that is a mixture of text and chunks of code. When
the document is processed by knitr, chunks of code will be
executed, and graphs or other results will be inserted into the final
document.
This sort of idea has been called “literate programming”.
knitr allows you to mix basically any type of text with
code from different programming languages, but we recommend that you use
R Markdown, which mixes Markdown with R. Markdown is a light-weight
mark-up language for creating web pages.
Creating an R Markdown file
Within RStudio, click File → New File → R Markdown and you’ll get a dialog box like this:
You can stick with the default (HTML output), but give it a title.
Basic components of R Markdown
The initial chunk of text (header) contains instructions for R to specify what kind of document will be created, and the options chosen. You can use the header to give your document a title, author, date, and tell it what type of output you want to produce. In this case, we’re creating an html document.
---
title: "Initial R Markdown document"
author: "Karl Broman"
date: "April 23, 2015"
output: html_document
---You can delete any of those fields if you don’t want them included. The double-quotes aren’t strictly necessary in this case. They’re mostly needed if you want to include a colon in the title.
RStudio creates the document with some example text to get you started. Note below that there are chunks like
```{r}
summary(cars)
```
These are chunks of R code that will be executed by
knitr and replaced by their results. More on this
later.
Markdown
Markdown is a system for writing web pages by marking up the text much as you would in an email rather than writing html code. The marked-up text gets converted to html, replacing the marks with the proper html code.
For now, let’s delete all of the stuff that’s there and write a bit of markdown.
You make things bold using two asterisks, like this:
**bold**, and you make things italics by using
underscores, like this: _italics_.
You can make a bulleted list by writing a list with hyphens or asterisks with a space between the list and other text, like this:
A list:
* bold with double-asterisks
* italics with underscores
* code-type font with backticksor like this:
A second list:
- bold with double-asterisks
- italics with underscores
- code-type font with backticksEach will appear as:
- bold with double-asterisks
- italics with underscores
- code-type font with backticks
You can use whatever method you prefer, but be consistent. This maintains the readability of your code.
You can make a numbered list by just using numbers. You can even use the same number over and over if you want:
1. bold with double-asterisks
1. italics with underscores
1. code-type font with backticksThis will appear as:
- bold with double-asterisks
- italics with underscores
- code-type font with backticks
You can make section headers of different sizes by initiating a line
with some number of # symbols:
# Title
## Main section
### Sub-section
#### Sub-sub sectionYou compile the R Markdown document to an html webpage by clicking the “Knit” button in the upper-left.
Challenge 1
Create a new R Markdown document. Delete all of the R code chunks and write a bit of Markdown (some sections, some italicized text, and an itemized list).
Convert the document to a webpage.
In RStudio, select File > New file > R Markdown…
Delete the placeholder text and add the following:
# Introduction
## Background on Data
This report uses the *gapminder* dataset, which has columns that include:
* country
* continent
* year
* lifeExp
* pop
* gdpPercap
## Background on Methods
Then click the ‘Knit’ button on the toolbar to generate an html document (webpage).
A bit more Markdown
You can make a hyperlink like this:
[Carpentries Home Page](https://carpentries.org/).
You can include an image file like this:

You can do subscripts (e.g., F2) with F~2~
and superscripts (e.g., F2) with F^2^.
If you know how to write equations in LaTeX, you can use
$ $ and $$ $$ to insert math equations, like
$E = mc^2$ and
$$y = \mu + \sum_{i=1}^p \beta_i x_i + \epsilon$$You can review Markdown syntax by navigating to the “Markdown Quick Reference” under the “Help” field in the toolbar at the top of RStudio.
R code chunks
The real power of Markdown comes from mixing markdown with chunks of code. This is R Markdown. When processed, the R code will be executed; if they produce figures, the figures will be inserted in the final document.
The main code chunks look like this:
```{r load_data}
gapminder
That is, you place a chunk of R code between ```{r
chunk_name} and ```. You should give each chunk a
unique name, as they will help you to fix errors and, if any graphs are
produced, the file names are based on the name of the code chunk that
produced them. You can create code chunks quickly in RStudio using the
shortcuts Ctrl+Alt+I on Windows and
Linux, or Cmd+Option+I on Mac.
Challenge
Challenge 2
Add code chunks to:
- Load the ggplot2 package
- Read the gapminder data
- Create a plot
```{r load-ggplot2}
library("ggplot2")
```
```{r read-gapminder-data}
gapminder
```{r make-plot}
plot(lifeExp ~ year, data = gapminder)
```
How things get compiled
When you press the “Knit” button, the R Markdown document is
processed by knitr
and a plain Markdown document is produced (as well as, potentially, a
set of figure files): the R code is executed and replaced by both the
input and the output; if figures are produced, links to those figures
are included.
The Markdown and figure documents are then processed by the tool pandoc, which converts the
Markdown file into an html file, with the figures embedded.
Chunk options
There are a variety of options to affect how the code chunks are treated. Here are some examples:
- Use
echo=FALSEto avoid having the code itself shown. - Use
results="hide"to avoid having any results printed. - Use
eval=FALSEto have the code shown but not evaluated. - Use
warning=FALSEandmessage=FALSEto hide any warnings or messages produced. - Use
fig.heightandfig.widthto control the size of the figures produced (in inches).
So you might write:
```{r load_libraries, echo=FALSE, message=FALSE}
library("dplyr")
library("ggplot2")
```
Often there will be particular options that you’ll want to use repeatedly; for this, you can set global chunk options, like so:
```{r global_options, echo=FALSE}
knitr::opts_chunk$set(fig.path="Figs/", message=FALSE, warning=FALSE,
echo=FALSE, results="hide", fig.width=11)
```
The fig.path option defines where the figures will be
saved. The / here is really important; without it, the
figures would be saved in the standard place but just with names that
begin with Figs.
If you have multiple R Markdown files in a common directory, you
might want to use fig.path to define separate prefixes for
the figure file names, like fig.path="Figs/cleaning-" and
fig.path="Figs/analysis-".
Challenge 3
Use chunk options to control the size of a figure and to hide the code.
```{r echo = FALSE, fig.width = 3}
plot(faithful)
```
You can review all of the R chunk options by navigating
to the “R Markdown Cheat Sheet” under the “Cheatsheets” section of the
“Help” field in the toolbar at the top of RStudio.
Inline R code
You can make every number in your report reproducible. Use
`r and ` for an in-line code chunk, like so:
`r round(some_value, 2)`. The code will be executed and
replaced with the value of the result.
Don’t let these in-line chunks get split across lines.
Perhaps precede the paragraph with a larger code chunk that does
calculations and defines variables, with include=FALSE for
that larger chunk (which is the same as echo=FALSE and
results="hide").
Rounding can produce differences in output in such situations. You
may want 2.0, but round(2.03, 1) will give
just 2.
The myround
function in the R/broman
package handles this.
Challenge 4
Try out a bit of in-line R code.
Here’s some inline code to determine that 2 + 2 = 4.
Other output options
You can also convert R Markdown to a PDF or a Word document. Click
the little triangle next to the “Knit” button to get a drop-down menu.
Or you could put pdf_document or word_document
in the initial header of the file.
Tip: Creating PDF documents
Creating .pdf documents may require installation of some extra
software. The R package tinytex provides some tools to help
make this process easier for R users. With tinytex
installed, run tinytex::install_tinytex() to install the
required software (you’ll only need to do this once) and then when you
knit to pdf tinytex will automatically detect and install
any additional LaTeX packages that are needed to produce the pdf
document. Visit the tinytex
website for more information.
Tip: Visual markdown editing in RStudio
RStudio versions 1.4 and later include visual markdown editing mode.
In visual editing mode, markdown expressions (like
**bold words**) are transformed to the formatted appearance
(bold words) as you type. This mode also includes a
toolbar at the top with basic formatting buttons, similar to what you
might see in common word processing software programs. You can turn
visual editing on and off by pressing the ![]() button in the top right corner of your R Markdown document.
button in the top right corner of your R Markdown document.
Resources
- Knitr in a knutshell tutorial
- Dynamic Documents with R and knitr (book)
- R Markdown documentation
- R Markdown cheat sheet
- Getting started with R Markdown
- R Markdown: The Definitive Guide (book by Rstudio team)
- Reproducible Reporting
- The Ecosystem of R Markdown
- Introducing Bookdown
- Mix reporting written in R Markdown with software written in R.
- Specify chunk options to control formatting.
- Use
knitrto convert these documents into PDF and other formats.
Content from Writing Good Software
Last updated on 2025-07-01 | Edit this page
Estimated time: 15 minutes
Overview
Questions
- How can I write software that other people can use?
Objectives
- Describe best practices for writing R and explain the justification for each.
Structure your project folder
Keep your project folder structured, organized and tidy, by creating
subfolders for your code files, manuals, data, binaries, output plots,
etc. It can be done completely manually, or with the help of RStudio’s
New Project functionality, or a designated package, such as
ProjectTemplate.
Tip: ProjectTemplate - a possible solution
One way to automate the management of projects is to install the
third-party package, ProjectTemplate. This package will set
up an ideal directory structure for project management. This is very
useful as it enables you to have your analysis pipeline/workflow
organised and structured. Together with the default RStudio project
functionality and Git you will be able to keep track of your work as
well as be able to share your work with collaborators.
- Install
ProjectTemplate. - Load the library
- Initialise the project:
R
install.packages("ProjectTemplate")
library("ProjectTemplate")
create.project("../my_project_2", merge.strategy = "allow.non.conflict")
For more information on ProjectTemplate and its functionality visit the home page ProjectTemplate
Make code readable
The most important part of writing code is making it readable and understandable. You want someone else to be able to pick up your code and be able to understand what it does: more often than not this someone will be you 6 months down the line, who will otherwise be cursing past-self.
Documentation: tell us what and why, not how
When you first start out, your comments will often describe what a command does, since you’re still learning yourself and it can help to clarify concepts and remind you later. However, these comments aren’t particularly useful later on when you don’t remember what problem your code is trying to solve. Try to also include comments that tell you why you’re solving a problem, and what problem that is. The how can come after that: it’s an implementation detail you ideally shouldn’t have to worry about.
Keep your code modular
Our recommendation is that you should separate your functions from
your analysis scripts, and store them in a separate file that you
source when you open the R session in your project. This
approach is nice because it leaves you with an uncluttered analysis
script, and a repository of useful functions that can be loaded into any
analysis script in your project. It also lets you group related
functions together easily.
Break down problem into bite size pieces
When you first start out, problem solving and function writing can be daunting tasks, and hard to separate from code inexperience. Try to break down your problem into digestible chunks and worry about the implementation details later: keep breaking down the problem into smaller and smaller functions until you reach a point where you can code a solution, and build back up from there.
Know that your code is doing the right thing
Make sure to test your functions!
Don’t repeat yourself
Functions enable easy reuse within a project. If you see blocks of similar lines of code through your project, those are usually candidates for being moved into functions.
If your calculations are performed through a series of functions, then the project becomes more modular and easier to change. This is especially the case for which a particular input always gives a particular output.
Remember to be stylish
Apply consistent style to your code.
- Keep your project folder structured, organized and tidy.
- Document what and why, not how.
- Break programs into short single-purpose functions.
- Write re-runnable tests.
- Don’t repeat yourself.
- Be consistent in naming, indentation, and other aspects of style.
